Detección de microRNAs extracelulares y su potencial como biomarcadores moleculares
FERMÍN MAR AGUILAR*, MARIO R. MORALES VALLARTA*, CRISTINA RODRÍGUEZ PADILLA*,
DIANA RESÉNDEZ PÉREZ*
CIENCIA UANL / AÑO 18, No. 71, ENERO-FEBRERO 2015
Los biomarcadores son moléculas que, al detectarse en tejidos biológicos, sirven como indicadores de las funciones normales o patológicas de un organismo. Un biomarcador ideal debe adaptarse a una serie de criterios en función de cómo va a utilizarse; debe ser accesible a través de métodos no invasivos, específico para la patología de interés y confiable para la detección de la enfermedad.
La mayoría de los biomarcadores detectables en sangre, en la actualidad, se basa en los niveles de proteínas específicas en la sangre. Aunque las proteínas son muy diversas y potencialmente muy informativas, diversos desafíos hacen que el descubrimiento y desarrollo de nuevos biomarcadores basados en proteínas sea una tarea costosa e intensiva. Estos desafíos incluyen la complejidad de la composición de proteínas en la sangre, la diversidad de las modificaciones postraduccionales y las dificultades en el desarrollo adecuado de agentes de detección de alta afinidad. (1)
Los microRNAs (miRNAs) son moléculas pequeñas de RNA con una longitud promedio de 22 nucleótidos, y con importantes funciones regulatorias a nivel postranscripcional, tanto en plantas como en animales. (2) Recientemente, se ha observado que estas moléculas reúnen muchas características de los buenos biomarcadores, por ejemplo, son estables en muchos fluidos corporales, y las secuencias de la mayoría se conservan entre especies diferentes. Además, el nivel de expresión de los miRNAs puede evaluarse con facilidad por varios métodos, entre los que se incluye la reacción en cadena de la polimerasa (PCR).
Otra ventaja de los miRNAs como biomarcadores es su expresión específica para los tejidos y representa muy bien su estado fisiológico. El perfil de expresión miRNAs definido se ha observado en patologías que van desde el cáncer hasta enfermedades parasitarias. (3-6)
Uno de los retos para combatir el cáncer de mama es encontrar un biomarcador mínimamente invasivo, sensible y específico que facilite la detección de esta enfermedad, el seguimiento del paciente y su respuesta al tratamiento.
Los estudios realizados sobre la expresión de los miRNAs en cáncer de mama indican que estas moléculas son específicas y, por sus características antes mencionadas, tienen potencial para ser excelentes biomarcadores y herramientas de pronóstico en esta patología. (7,8.)
En cuanto a las enfermedades parasitarias, se postula que los miRNAs podrían influenciar la expresión génica en su hospedero para facilitar la infección y controlar su respuesta inmune. (9) Algunos de los miRNAs identificados en parásitos han mostrado potencial para la identificación de una especie específica. (10) Sin embargo, la identificación de miRNAs en estos organismos sigue siento bastante limitada, y se necesitan ensayos de secuenciación para caracterizar estas moléculas y descubrir nuevos biomarcadores.
En el presente trabajo se muestra que el perfil de expresión de miRNAs permite distinguir con alta sensibilidad, especificidad y eficiencia entre pacientes con cáncer mamario (CM) e individuos sanos. La combinación de tres miRNAs usados al mismo tiempo como un solo grupo eleva significativamente la eficiencia de la prueba en cáncer mamario. La alta estabilidad de los miRNAs en suero sanguíneo permitió su detección, y nos permite postular que tienen potencial para convertirse en una herramienta de diagnóstico no invasiva. Adicionalmente, en nuestro laboratorio se secuenciaron por primera vez los miRNAs de Entamoeba histolytica, lo que permitió detectar 199 miRNAs no descritos anteriormente, y este hallazgo fue validado mediante la determinación de la expresión de los miRNAs con microarreglos y por RT-PCR tiempo real. Este hallazgo constituye el primero en su tipo, y supone el inicio de la búsqueda de la función de los miRNAs de E. histolytica.
METODOLOGÍA
Las muestras de tejido y suero sanguíneo de pacientes con cáncer de mama, así como de pacientes con amibiasis, se almacenaron a -70°C hasta su utilización.
Para la extracción de los RNAs totales de las muestras de tejido de CM, de los sueros sanguíneos y trofozoítos de E. histolytica HM1-IMSS, se utilizó el Kit miRNeasy, con base en el protocolo del fabricante.
PCR tiempo real
Las muestras de tejido y suero sanguíneo de pacientes con cáncer mamario fueron amplificadas por PCR tiempo real, con sondas Taqman, de acuerdo a las instrucciones del proveedor. La cuantificación fue realizada por medio del método de 2-˜Ct, con tejido y suero sanguíneo de mujeres sanas como calibrador.
Secuenciación de nueva generación (NGS)
La biblioteca de DNAc fue secuenciada en el equipo Illumina Gallx, a partir de las instrucciones del proveedor. Las lecturas de la secuenciación sin procesar se obtuvieron con el software de Illumina Sequencing Control Studio versión 2.8 (SCS v2.8), seguido del análisis de imagen de secuenciación en tiempo real y lectura automática de nucleótidos de Illumina Real-Time Analysis versión 1.8.70 (RTA v1.8.70). El análisis de las secuencias obtenidas se realizó con el programa ACGT101-miR v4.2 (LC Sciences). (11-13)
RESULTADOS
En el presente estudio, se determinó la expresión diferencial de miR-10b, miR-21, miR-125b, miR-145, miR-155 y miR-191 en 50 pacientes con cáncer de mama, con respecto a la expresión de estos miRNAs en 20 tejidos normales de mama. Después del análisis estadístico, se seleccionaron los miRNAs que presentaron desregulación estadísticamente significativa (miR-21, miR-125b y miR-191), para determinar sus niveles de expresión. La expresión relativa de miR-21 y miR-191 fue más alta en el tejido de cáncer de mama, con respecto al tejido normal, mientras que la expresión de miR-125b presentó menor expresión. La sobreexpresión de miR-21 se elevó hasta 20 veces en 46 de las 50 muestras; en miR-191 la sobreexpresión se elevó hasta 200 veces en 49 de las 50 muestras, en contraste con miR-125b que mostró subexpresión de hasta cuatro veces menos, con respecto al tejido normal en 48 de las 50 muestras (figura 1).
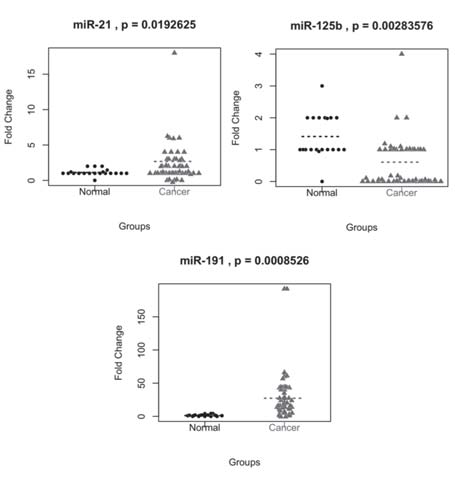
Fig. 1. Niveles de expresión relativa de miR-21, miR125b y miR-191 en cáncer
de mama y tejido sano. Los niveles de expresión relativa y valores estadísticos
(p) de miR-21 (p=0.01), miR-125b (p=0.002) y miR-191 (p=0.0008), en
pacientes con cáncer de mama comparados con tejido de mama sano. La línea
punteada representa la media del nivel de expresión para cada miRNA.
Los valores de p de los 3 miRNAs (p= 0.019 para miR-21, p=0.002 para miR-125b y p= 0.0008 para miR-191) señalan que es posible discernir entre una muestra de cáncer de mama comparada con tejido normal sólo evaluando la expresión relativa. A partir de los valores de expresión relativa, se construyeron curvas ROC para cada uno de los miRNAs analizados; y se realizó el cálculo del área bajo la curva ROC (AROC; figura 2). Con los valores de AROC se calcularon sensibilidad, especificidad, valor de predicción positiva (VPP), valor de predicción negativa (VPN) y exactitud. Los valores de sensibilidad y especificidad fueron de 75 y 60% para miR-21, 79 y 88% para miR-125b y 93 y 95% para miR-191, respectivamente. Adicionalmente, se calcularon estos valores para la combinación de 2 miRNAs, siendo la sensibilidad y especificidad de 100 y 94% para la combinación de miR-125b/miR-191, y 92 y 100% para la combinación de miR-21/miR-191, respectivamente. Los valores de VPP y VPN fueron de 100 y 88% para la combinación de miR-21/miR-191, y 96 y 100% para la combinación de miR-125b/miR-191, respectivamente. Asimismo, los valores de exactitud de la prueba fueron de 98% para la combinación miR-125b/miR-191, y 95% para la combinación miR-21/miR-191. Los resultados muestran que la combinación de dos miRNAs (miR-125b/miR-191 y miR-21/miR-191) presentaron mayor capacidad para diferenciar entre tejido de cáncer de mama y tejido sano. (14)
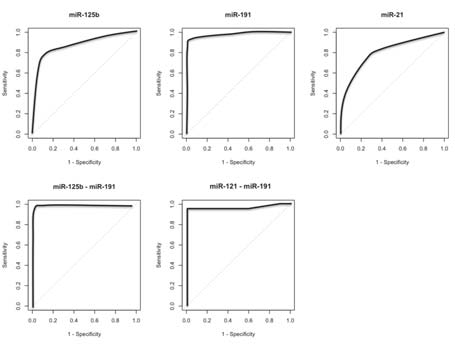
Fig. 2. Área bajo la curva ROC (AROC) para miR-21, miR-125b, miR-191 y
las combinaciones de miRs. La curva muestra la sensibilidad y especificidad
para miR-21, miR-125b, miR-191, miR-125b/miR-191 y miR-21/miR-191.
Con base en estos resultados, posteriormente se cuantificaron los niveles de expresión de miR-10b, miR-21, miR-125b, miR-45, miR-155, miR-186, miR-191 y miR-382 en suero sanguíneo de 65 pacientes con cáncer de mama y diez controles sanos, para determinar si existen patrones diferenciales entre ambos casos por RT-PCR tiempo real. Los resultados de los niveles de expresión manifestaron que en 42 muestras de sueros de pacientes con CM amplificadas satisfactoriamente para miR-10b, su totalidad presentó una sobreexpresión con un rango que va desde 1.5 a 11, 569.2 veces más que el promedio de expresión para las pacientes sanas. De 59 muestras amplificadas satisfactoriamente para miR-21, 56 verificaron la sobreexpresión con rangos de 1.1- 24, 423.8 veces más a los controles sanos, mientras que el resto presentó subexpresión con un rango de 5.6-50 veces menos al de los controles sanos. Las 40 muestras amplificadas satisfactoriamente para miR-125b se sobreexpresaron de 2.7 a 115, 777.9 veces más, en comparación a las controles sanos.
Las 18 muestras amplificadas para miR-145 se sobreexpresaron de 1.6 a 56, 423.2 veces más. De las 41 muestras amplificadas para miR-155, 40 se sobreexpresaron de 1.8 a 129, 716.3 veces más, mientras que una presentó subexpresión de 1.6 veces menos comparada con los controles sanos. De las 33 muestras amplificadas para miR-186, 31 presentaron sobreexpresión de 1.07 a 17, 817.41 veces más, y dos mostraron subexpresión de 1.6 a 2.3 veces menor que los controles sanos. De 25 muestras para miR- 191, 21 presentan sobreexpresión de 1.4 a 4, 138.8 veces mayor, y cuatro se subexpresan de 1.1 a 50 veces menos que en pacientes sanas. Por último, las 40 muestras amplificadas para miR-382 presentaron sobreexpresión de 2.43 a 1, 948.3 veces más que en pacientes sanas.
En la figura 3, se muestra que la concentración de ocho miRNAs fue significativamente mayor en el suero de los pacientes con CM que en el de los pacientes sanos (p < 0.001). El análisis de la curva ROC, llevada a cabo para evaluar el valor diagnóstico de los ocho miRNAs en suero, reveló el potencial de tres de ellos (miR-145, miR- 155 y miR-382) como biomarcadores valiosos para la distinción de pacientes con CM de los pacientes sanos.
Se calculó el valor límite óptimo de 59.22 para una máxima sensibilidad y especificidad de miR-10b, en el que los valores fueron de 83.30 y 100.00%, respectivamente. El valor límite de 6.48 para miR-21 mostró la sensibilidad y especificidad de 94.40 y 80.00%; en el valor límite de 8.46 para miR-125b, la sensibilidad y especificidad fueron de 88.90 y 80.00%. En el valor límite de 15.93 para miR-145, la sensibilidad y especificidad fueron de 94.40 y 100.00%, y en el valor límite de 7.92 para miR-155, la sensibilidad y especificidad fueron de 94.40 y 100.00%.
En el valor límite de 9.07 para miR-186, la sensibilidad y especificidad fueron de 83.30 y 90.00%, y en el valor límite de 11.59 para miR-191, la sensibilidad y especificidad fueron de 72.20 y 90.00%. Por último, en el valor límite de 4.85 para miR-382, la sensibilidad y especificidad fueron de 94.40 y 90.00% (figura 3).
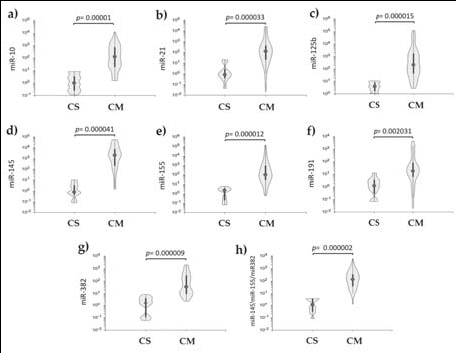
Fig. 3. Niveles relativos de expresión de los miRNAs en el suero de pacientes
con CM. a) miR-10b, b) miR-21, c) miR-125b, d) miR-145, e) miR-155, f)
miR-191, g) miR-382, h) miR-145/ miR-155/ miR-382 en suero de pacientes
con cáncer de mama (CM) y controles sanos (CS). Los valores p para todos
los microRNAs fueron significativos (p < 0.001).
Para elevar el valor diagnóstico del ensayo se realizó la combinación de las curvas ROC de miR-145, miR-155 y miR-82. El radio de combinación de estos tres miRNAs logró los mayores valores de AUC de 0.9878 (95% CI: 0.9065 a 0.9985); y en el límite óptimo de 10.07, la sensibilidad y especificidad fueron de 97.60 y 100.00%, respectivamente (figura 4). Finalmente, la eficiencia de la prueba diagnóstica, definida como el porcentaje de sujetos diferenciados correctamente como enfermos de los sujetos sanos fue de 89.3% para miR-10b, 89.3% para miR-21, 89.3% para miR-125b, 96.40% para miR-145, 96.40% para miR-155, 85.7% para miR-186, 78.60% para miR-191, 92.90% para miR-382, y de 98% para la combinación de miR-145/miR-155/ miR-382. Estos resultados indican que el análisis de tres miRNAs simultáneamente tiene un mayor potencial diagnóstico para la detección de cáncer de mama, en comparación de la detección de un solo miRNA como biomarcador.
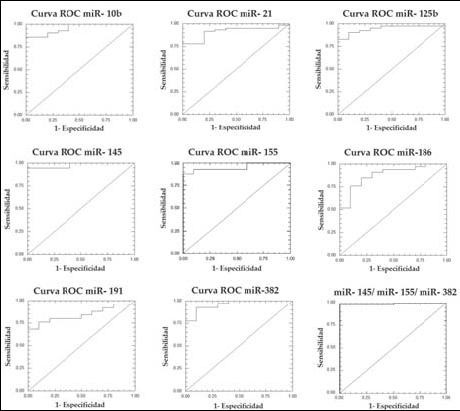
Fig. 4. Área bajo la curva ROC de miR-10b, miR-21, miR-125b, miR-145,
miR-155, miR-186, miR-191, miR-382 y miR-145/miR-155/miR-382. Cada
miRNA por separado mostró un menor valor de sensibilidad y especificidad
que la combinación de miR-145/miR-155/miR-382.
Los niveles séricos de los miR-10b, miR-21, miR-125b, miR-145, miR-155, miR-186, miR-191 y miR-382 se analizaron en pacientes con CM en diferentes estadios TNM de la enfermedad, para determinar si estos miRNAs podrían diferenciar entre estadios. Los resultados no mostraron diferencia significativa entre los niveles séricos de los ocho miRNAs en los cuatro estadios analizados de la enfermedad. (15,16) Por otro lado, también se determinó la presencia de los microRNAs en trofozoítos de Entamoeba histolytica HM1-IMSS, mediante secuenciación de nueva generación (NGS).
De la reacción de secuenciación se obtuvieron en total 16, 688, 748 lecturas de secuencias sin procesar; de éstas fueron removidas las secuencias que no contenían el adaptador utilizado para la secuenciación, resultando en 10, 620, 021 lecturas, de las cuales se eliminaron aquéllas que tenían menos de 15 nucleótidos de longitud. Finalmente, 7, 604 secuencias fueron consideradas como contaminantes por lo que, al eliminarse, resultaron 5, 239, 324 secuencias nucleotídicas en total, que se consideraron como lecturas asignables. Estas lecturas asignables se clasificaron en grupos, y las de mayor importancia fueron las que se pudieron alinear al genoma de E. histolytica, además, estas secuencias extendidas fueron alineadas al genoma de E. histolytica para determinar el potencial de formar horquillas (hairpins). En este grupo se encontraron 199 miRNAs diferentes, con 21 nucleótidos de longitud en promedio.
Predicción de genes blanco regulados por miRNAs en Entamoeba histolytica
Para determinar la función probable de los miRNAs de la amiba, se realizó la predicción de genes blanco con el programa miRanda, (17) el cual es un algoritmo computacional de código abierto con una tasa conocida de falsos descubrimientos. (17,18) Para el genoma de E. histolytica existen 8, 201 genes codificantes anotados, 90% de los cuales no tiene una 3’UTR anotada. En 10% de los genes de E. histolytica con una región 3’UTR anotada, ésta es de longitud variable. En este trabajo utilizamos 340 genes con 3’UTR y, para realizar un análisis más exacto, consideramos todas las regiones 3’UTR que tenían la misma longitud. Para uniformizar esta medida, se calculó la media de la longitud de las regiones 3’UTR, y se le sumaron dos desviaciones estándar (media observada= 140; desviación estándar= 180).
De esta manera, se extrajeron de la base de datos secuencias de 500 nucleótidos de 3’UTR, más 30 nucleótidos de la parte final de la secuencia codificante. La energía libre mínima (MFE) proporciona una idea de la estabilidad de la estructura secundaria del miRNA. Wei y colaboradores han usado este parámetro en -14 kCal/mol, (19) mientras que Huang et al. han usado en -25 kCal/mol. (20) Para este trabajo, se eligió el valor intermedio de MFE, y se fijó en -18 kCal/mol para llevar a cabo el análisis en el algoritmo miRanda. Se obtuvieron 66 posibles genes blanco, 32 con genes hipotéticos y 34 con genes con función conocida. Estos resultados sugieren que los miRNAs presentes en E. histolytica podrían regular de uno a ocho genes diferentes. (21)
DISCUSIÓN
En el presente trabajo se analizó el perfil de expresión de nueve miRNAs en tejido mamario; los miRNAs se seleccionaron debido al análisis de microarreglos que reveló un patrón de expresión diferencial en tejido canceroso contra tejido normal. Elegimos miR-10b, miR-125 y miR-145, porque se encuentran menos expresados en tejido canceroso con respecto al tejido normal; a miR-21 y miR-155, porque se encuentran sobreexpresados en tejido maligno. Asimismo, miR-191, miR-212 y miR-213 se eligieron debido a que pueden predecir el grado de invasividad del tumor.
El análisis estadístico realizado a los resultados obtenidos demuestra que miR-10, miR-21, miR- 125b, miR-132, miR-45, miR-212 y miR-213 evidenciaron diferencia significativa, siendo miR-10, miR-125b, miR-21 y miR-145 los mejores miRNAs para el diagnóstico diferencial entre tejido sano y tejido canceroso. Los miRNAs restantes no mostraron diferencia significativa, aunque el análisis de un mayor número de muestras permitirá precisar estas cifras. La combinación de dos miRNAs (mir-21/miR-191 y miR-125b/miR-191) mostró un aumento de la sensibilidad y especificidad del ensayo, por lo que se considera que son los mejores candidatos para utilizarse como biomarcadores. Estos resultados obtenidos apoyan la posibilidad de utilizar a los miRNAs analizados como posibles biomarcadores moleculares, ya que pueden discriminar entre tejido normal y tejido canceroso en la mayoría de los casos analizados.
Recientemente, se ha observado que los miRNAs extracelulares que circulan en corriente sanguínea pueden utilizarse como posibles biomarcadores para el diagnóstico de muchas enfermedades, incluyendo el cáncer. (22-26) A la fecha se han realizado varios estudios para medir los niveles de expresión de los miRNAs en cáncer de mama, tanto en tejido tumoral como en suero y plasma. (26,27) Los resultados obtenidos en nuestro laboratorio con el análisis de la expresión de miRNAs en tejido de cáncer mama fue el punto de partida para analizar el perfil de expresión de estos miRNAs en suero de pacientes con cáncer de mama.
En la búsqueda del posible establecimiento de nuevos biomarcadores, analizamos el perfil de expresión de seis miRNAs desregulados en tejido (miR- 10b, miR-21, miR-125b, miR-145, miR-155 y miR-191), mientras que miR-186 y miR-382 se incluyeron en el análisis, tomando en cuenta los resultados obtenidos en ensayos con placas TLDA (Taqman Low Density Array) realizados en nuestro laboratorio. El análisis se realizó en suero de 65 pacientes con cáncer de mama, y diez mujeres sanas (controles). La sensibilidad y especificidad para los ocho miRNAs analizados con un límite óptimo fueron de 83.30 y 100.00% para miR-10b, 94.40; 80.00% para miR-21, 88.90 y 80.00 para miR-125b, 94.40 y 100.00% para miR-145, 94.40 y 100.00% para miR-155, 83.30 y 90.00% para miR-186, 72.20 y 90.00% para miR-191 y 94.40 y 90.00% para miR-382. Los resultados obtenidos concuerdan con otros estudios que discriminan sujetos con cáncer y sujetos sanos. (14,28,29) Además, se detectó que la combinación de tres miRNAs (miR-145, miR-155 y miR-382) tiene una mejor sensibilidad (97.6%), especificidad (100%) y eficiencia (98%) que cualquier otro miRNA analizado por separado.
Por otro lado, como los miRNAs están involucrados en muchos procesos de regulación en la célula es posible que estén involucrados en los mecanismos que utilizan parásitos como E. histolytica. Sin embargo, hasta la fecha no ha sido reportada la existencia de miRNAs en Entamoeba histolytica. Por ello, se planteó el objetivo de determinar la presencia de miRNAs en E. histolytica. Para llevar a cabo este objetivo, se utilizó la secuenciación de nueva generación, la cual ofrece la ventaja de cuantificar la abundancia absoluta de los miRNAs, y además permite el descubrimiento de nuevos miRNAs. Se obtuvieron 16 millones de secuencias, de las cuales sólo poco más de 5 millones fueron lecturas de alta calidad en el genoma de E. histolytica. Se encontró que sólo 0.5% de las lecturas no fueron redundantes y se alinearon de manera perfecta al genoma de E. histolytica, y estas secuencias son candidatas a ser nuevos miRNAs, debido a que su secuencia tiene el potencial de formar una estructura de horquilla, la cual es característica de los precursores de los miRNAs.
En este trabajo se identificaron por primera vez 199 miRNAs pertenecientes a E. histolytica, esto de acuerdo a los criterios para distinguir miRNAs de otros tipos de RNAs pequeños, (30-32) a partir del esquema bioinformático propuesto en el algoritmo ACGT101-miR v4.2.11,13 Nuestros resultados no mostraron ninguna similitud con miRNAs conocidos de plantas y animales. Estos resultados son consistentes con los hallazgos observados en el alga verde unicelular Chlamydomonas reinhardtii, la cual también posee miRNAs, pero ninguno presenta similitud con los miRNAs reportados hasta el momento. (33). Este resultado sugiere que los de organismos unicelulares representan una nueva clase de miRNAs que merecen estudiarse con amplitud. Adicionalmente, la falta de miRNAs universalmente conservados entre plantas, animales y algas verdes sugiere que los genes de miRNAs pudieron haber evolucionado independientemente en los linajes que conducen a estos grupos. (33) Por lo tanto, la evolución de E. histolytica podría ser similar a la de las algas verdes.
Recientemente, se ha acumulado evidencia sobre la función de los miRNAs en la interacción hospedero-patógeno y la regulación de la respuesta inmune contra agentes infecciosos. Debido a que los parásitos han coevolucionado con sus hospederos para el establecimiento de infecciones crónicas y el desarrollo de mecanismos de evasión, se ha postulado la hipótesis de que los parásitos usan estos mecanismos para evadir la respuesta inmune del hospedero, y tomando en cuenta que los miRNAs no generan una respuesta inmune, pueden transportarse libremente vía exosomas, y regular los transcritos de las células del hospedero. Sin embargo, a la fecha no se ha establecido una interacción de esta naturaleza.(34,35) El estudio de los miRNAs presentes en parásitos, su expresión y transporte hacia las células del hospedero servirá como base del establecimiento de nuevos biomarcadores y terapias para las enfermedades producidas por parásitos.
La búsqueda de biomarcadores informativos no sólo es clave para entender los procesos fisiopatológicos de las enfermedades, también es fundamental para el desarrollo terapéutico, por lo que ha sido uno de los principales enfoques de la investigación biomédica en las últimas dos décadas. A pesar de la identificación de nuevos biomarcadores basados en proteínas, muy pocos pasan procesos realmente rigurosos de validación. Los miRNAs poseen muchas propiedades deseables en comparación con biomarcadores de proteínas; sin embargo, requieren los mismos procesos de validación para demostrar su especificidad y selectividad. Los desafíos fundamentales del desarrollo de biomarcadores basados en miRNAs son las cuestiones relacionadas con su medición exacta. La creación de un proceso de estandarización para la preparación de muestras y el desarrollo de un método más preciso para evaluar la calidad y cantidad de los miRNAs son los desafíos del presente. Una vez resueltos los métodos de diagnóstico basados en miRNAs, podrían ser una realidad en el laboratorio clínico.
RESUMEN
Los microRNAs (miRNAs) son moléculas de RNA de 18 a 24 nucleótidos de longitud involucrados en la regulación de la expresión génica a nivel postranscripcional en la célula. En este trabajo analizamos la expresión de miRNAs en muestras de tejido y suero de pacientes con cáncer mamario, y este perfil permite distinguir pacientes con cáncer de mama y mujeres sanas con alta sensibilidad, especificidad y eficiencia. La combinación de dos miRNAs para tejido y tres para suero, usados en conjunto como un solo grupo, eleva significativamente la eficiencia de la prueba, y demuestra que los miRNAs tienen gran potencial como nuevos biomarcadores no invasivos en cáncer de mama. Adicionalmente, se realizó la búsqueda de miRNAs en el protozoario Entamoeba histolytica, por secuenciación profunda, lo que permitió realizar la primera descripción de miRNAs en este parásito. Se identificaron 199 miRNAs exclusivos para este parásito, que servirán de base para el estudio de la regulación génica en E. histolytica y el establecimiento de nuevos biomarcadores para la amibiasis.
Palabras clave: miRNAs, Cáncer de mama, E. histolytica.
ABSTRACT
MicroRNAs (miRNAs) are 18 to 24 nucleotide-long RNA molecules responsible for the regulation of gene expression at the post-transcriptional level in the cell. In this work, we analyzed the expression of miRNAs in tissue samples and serum of patients with breast cancer and we found that miRNA expression patterns distinguish patients with breast cancer from healthy women with high sensitivity, specificity, and efficiency. The combination of 2 and 3 miRNAs used as a group in tissue and serum respectively, significantly increases the efficiency of the test and shows that miRNAs have potential as novel noninvasive biomarkers in breast cancer detection. Additionally, miRNAs were detected by deep sequence in the protozoan parasite Entamoeba histolytica, which allowed the first description of miRNAs in this parasite. We identified 199 new miRNAs in E. histolytica, which are the base for the study of gene regulation and the establishment of new biomarkers for amoebiasis.
Keywords: MiRNAs, Breast Cancer, E. histolytica.
AGRADECIMIENTOS
Los autores agradecen a los Cuerpos Académicos de Inmunología y Biología Celular y Genética de la FCB, por su colaboración multidisciplinaria y apoyo al presente trabajo. Asimismo, a Alejandra Arreola Triana, por la revisión del manuscrito, y al apoyo financiero de los proyectos Promep:103.5/07/2523 y 103.5/08/4285. Paicyt: GCN 040-10.
*Universidad Autónoma de Nuevo León, FCB.
dianaresendezpr@uanl.edu.mx
——————
El presente artículo está basado en la investigación “Detección de microRNAs extracelulares y su potencial como biomarcadores moleculares”, galardonado con el Premio de Investigación UANL 2014 en la categoría de Ciencias Naturales, otorgado en sesión solemne del Consejo Universitario de la UANL, en agosto de 2014.
Referencias
1. Mayeux R. (2004). Biomarkers: potential uses and limitations. NeuroRx 1: 182-188.
2. Bartel D.P. (2004). MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 116: 281-297.
3. Cheng G. (2014). Circulating miRNAs: Roles in cancer diagnosis, prognosis and therapy. Adv Drug Deliv Rev.
4. Tritten L., Burkman E., Moorhead A., Satti M., Geary ,J. et al. (2014). Detection of circulating parasite-derived microRNAs in filarial infections. PLoS Negl Trop Dis 8: e2971.
5. Gallo A., Tandon M., Alevizos I., Illei G.G. (2012). The majority of microRNAs detectable in serum and saliva is concentrated in exosomes. PLoS One 7: e30679.
6. Wang Y., Zheng D., Tan Q., Wang M.X., Gu L.Q. (2011). Nanopore-based detection of circulating microRNAs in lung cancer patients. Nat Nanotechnol 6: 668-674.
7. Iorio M.V., Ferracin M., Liu C.G., Veronese A., Spizzo R., et al. (2005). MicroRNA gene expression deregulation in human breast cancer. Cancer Res 65: 7065-7070.
8. Mattie M.D., Benz C.C., Bowers J., Sensinger K., Wong L., et al. (2006). Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer 5: 24.
9. Chen M.X., Ai L., Xu M.J., Chen S.H., Zhang Y.N., et al. (2011). Identification and characterization of microRNAs in Trichinella spiralis by comparison with Brugia malayi and Caenorhabditis elegans. Parasitol Res 109: 553-558.
10. Hao L., Cai P., Jiang N., Wang H., Chen Q. (2010). Identification and characterization of microRNAs and endogenous siRNAs in Schistosoma japonicum. BMC Genomics 11: 55.
11. Li M., Xia Y., Gu Y., Zhang K., Lang Q., et al. (2010). MicroRNAome of porcine pre- and postnatal development. PLoS One 5: e11541.
12. Meyer C., Grey F., Kreklywich C.N., Andoh T.F., Tirabassi R.S., et al. (2011). Cytomegalovirus microRNA expression is tissue specific and is associated with persistence. J Virol 85: 378-389.
13. Wei Z., Liu X., Feng T., Chang Y. (2011). Novel and conserved micrornas in Dalian purple urchin (Strongylocentrotus nudus) identified by next generation sequencing. Int J Biol Sci 7: 180-192.
14. Mar Aguilar F., Luna Aguirre C.M., Moreno Rocha J.C., Araiza Chávez J., Treviño V. et al. (2013). Differential expression of miR-21, miR-125b and miR-191 in breast cancer tissue. Asia Pac J Clin Oncol 9: 53-59.
15. Mar Aguilar F, Mendoza Ramírez J.A., Malagón Santiago I., Espino Silva P.K., Santuario Facio S.K., et al. (2013). Serum circulating microRNA profiling for identification of potential breast cancer biomarkers. Dis Markers 34: 163-169.
16. Mar Aguilar F., Rodríguez Padilla C., Reséndez Pérez D. (2014). Use of serum-circulating miRNA profiling for the identification of breast cancer biomarkers. Methods Mol Biol 1165: 71-80.
17. Enright A.J., John B., Gaul U., Tuschl T., Sander C. et al. (2003). MicroRNA targets in Drosophila. Genome Biol5: R1.
18. John B., Enright A.J., Aravin A., Tuschl T., Sander C., et al. (2004). Human MicroRNA targets. PLoS Biol 2: e363.
19. Wei Y., Chen S., Yang P., Ma Z., Kang L. (2009). Characterization and comparative profiling of the small RNA transcriptomes in two phases of locust. Genome Biol 10: R6.
20. Huang J., Hao P., Chen H., Hu W., Yan Q., et al. (2009). Genome-wide identification of Schistosoma japonicum microRNAs using a deep-sequencing approach. PLoS One 4: e8206.
21. Mar Aguilar F., Treviño V., Salinas Hernández J.E., Tamez Guerrero M.M., Barrón González M.P., et al. (2013). Identification and Characterization of microRNAS from Entamoeba histolytica HM1-IMSS. PLoS One 8: e68202.
22. Mitchell P.S., Parkin R.K., Kroh E.M., Fritz B.R., Wyman S.K., et al. (2008). Circulating microRNAs as stable bloodbased markers for cancer detection. Proc Natl Acad Sci U S A 105: 10513-10518.
23. Gilad S., Meiri E., Yogev Y., Benjamin S., Lebanony D., et al. (2008). Serum microRNAs are promising novel biomarkers. PLoS One 3: e3148.
24. Chen X., Ba Y., Ma L., Cai X., Yin Y., et al. (2008). Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res 18: 997-1006.
25. Resnick K.E., Alder H., Hagan J.P., Richardson D.L., Croce C.M., et al. (2009). The detection of differentially expressed microRNAs from the serum of ovarian cancer patients using a novel real-time PCR platform. Gynecol Oncol 112: 55-59.
26. Wang K., Zhang S., Marzolf B., Troisch P., Brightman A., et al. (2009). Circulating microRNAs, potential biomarkers for drug-induced liver injury. Proc Natl Acad Sci U S A 106: 4402-4407.
27. Cookson V.J., Bentley M.A., Hogan B.V., Horgan K., Hayward B.E., et al. (2012). Circulating microRNA profiles reflect the presence of breast tumours but not the profiles of microRNAs within the tumours. Cell Oncol (Dordr) 35: 301-308.
28. Wang S., Wang L., Bayaxi N., Li J., Verhaegh W., et al. (2013). A microRNA panel to discriminate carcinomas from high-grade intraepithelial neoplasms in colonoscopy biopsy tissue. Gut 62: 280-289.
29. Chen Z.H., Zhang G.L., Li H.R., Luo J.D., Li Z.X., et al. (2012). A panel of five circulating microRNAs as potential biomarkers for prostate cancer. Prostate.
30. Ruby J.G., Jan C., Player C., Axtell M.J., Lee W., et al. (2006). Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs in C. elegans. Cell 127: 1193-1207.
31. Grimson A., Srivastava M., Fahey B., Woodcroft B.J., Chiang H.R., et al. (2008). Early origins and evolution of microRNAs and Piwi-interacting RNAs in animals. Nature 455: 1193-1197.
32. Ruby J.G., Stark A., Johnston W.K., Kellis M., Bartel D.P., et al. (2007). Evolution, biogenesis, expression, and target predictions of a substantially expanded set of Drosophila microRNAs. Genome Res 17: 1850-1864.
33. Zhao T., Li G., Mi S., Li S., Hannon G.J. et al. (2007). A complex system of small RNAs in the unicellular green alga Chlamydomonas reinhardtii. Genes Dev 21: 1190- 1203.
34. Umbach J.L., Nagel M.A., Cohrs R.J., Gilden D.H., Cullen B.R. (2009). Analysis of human alphaherpesvirus microRNA expression in latently infected human trigeminal ganglia. J Virol 83: 10677-10683.
35. Hu G., Zhou R., Liu J., Gong A.Y., Chen X.M. (2010). MicroRNA-98 and let-7 regulate expression of suppressor of cytokine signaling 4 in biliary epithelial cells in response to Cryptosporidium parvum infection. J Infect Dis 202: 125-135.
Recibido: 18/07/14
Aceptado: 18/08/14