Un giro en la Biología: explorando los ARNs circulares y su impacto
 Jossephlyn Hernández Alcántara* ORCID: 0000-0003-4833-594,
Jossephlyn Hernández Alcántara* ORCID: 0000-0003-4833-594,
Adriana Domínguez Vázquez* ORCID: 0000-0003-3186-4365
Cynthia Gabriela Sámano Salazar* ORCID: 0000-0002-8909-9582
CIENCIA UANL / AÑO 27, No.124, marzo-abril 2024
Los avances tecnológicos en la secuenciación genómica han revelado la organización y contenido del genoma, el cual alberga una mayor cantidad de genes que codifican para moléculas de ácido ribonucleico (ARN) en comparación con los que lo hacen para proteínas. De los 63,494 genes presentes en el genoma, sólo 19,969 codifican las segundas, mientras que 27,499 lo hacen en el primero (Human, s.a.; Ezkurdia et al., 2014; Nurk et al., 2022).
En la actualidad, el ARN por definición es una molécula de cadena sencilla presente en todas las células vivas. Dicha molécula, de gran relevancia biológica y multifacética, juega un papel central y moderador en la expresión génica. Traduce los mensajes contenidos en los genes del ácido desoxirribonucleico (ADN), lo que lleva a la síntesis de las proteínas esenciales en el funcionamiento celular (Dai, Zhang y Zaleta-Rivera, 2020). Su importancia es tan destacada que teorías, entre ellas la del “mundo del ARN”, han sugerido que la vida en la Tierra pudo haber surgido gracias a la versatilidad de las moléculas de ARN. Aunque esto se mantiene en hipótesis, constituye uno de los pilares fundamentales en la comprensión del origen de la vida (Saito, 2022).
 Es posible reconocer dos categorías principales de ARN: los codificantes y los no codificantes. Los primeros (ARNc) engloban a los mensajeros (ARNm), destinados a ser traducidos para la síntesis de proteínas. Por otro lado, los segundos (ARNnc) no se traducen en éstas. Sin embargo, su falta de producción proteica no debe subestimarse, ya que no implica que carezcan de relevancia (Zhang et al., 2019). Al día de hoy, aproximadamente 99% del total de ARN se compone de ARNnc. A medida que se han ido descubriendo más ARNnc con el tiempo, ha surgido una reevaluación de su misión en el mantenimiento de la homeostasis celular (Statello et al., 2021). Ahora se reconoce que son elementos activos, versátiles y altamente prevalentes en las células. Cumplen un papel fundamental en la regulación de la expresión génica y en una variedad de procesos biológicos y patológicos (Panni et al., 2020). El mundo del ARN es vasto y diverso, pudiera compararse con una familia de distintos miembros que podrían ser diferenciados y categorizados según su tamaño y labor.
Es posible reconocer dos categorías principales de ARN: los codificantes y los no codificantes. Los primeros (ARNc) engloban a los mensajeros (ARNm), destinados a ser traducidos para la síntesis de proteínas. Por otro lado, los segundos (ARNnc) no se traducen en éstas. Sin embargo, su falta de producción proteica no debe subestimarse, ya que no implica que carezcan de relevancia (Zhang et al., 2019). Al día de hoy, aproximadamente 99% del total de ARN se compone de ARNnc. A medida que se han ido descubriendo más ARNnc con el tiempo, ha surgido una reevaluación de su misión en el mantenimiento de la homeostasis celular (Statello et al., 2021). Ahora se reconoce que son elementos activos, versátiles y altamente prevalentes en las células. Cumplen un papel fundamental en la regulación de la expresión génica y en una variedad de procesos biológicos y patológicos (Panni et al., 2020). El mundo del ARN es vasto y diverso, pudiera compararse con una familia de distintos miembros que podrían ser diferenciados y categorizados según su tamaño y labor.
Según su tamaño, es posible dividirlos en pequeños (ARNncp) y largos (ARNncl). Estas moléculas se encuentran presentes en diversas especies animales y desempeñan un papel fundamental en procesos fisiológicos y patológicos (Hombach y Kretz, 2016). De acuerdo a su actividad existen ARN no codificantes constitutivos que participan en procesos celulares esenciales (figura 1a). Por ejemplo, los ribosomales (ARNr) y los de transferencia (ARNt) contribuyen al proceso de traducción en la síntesis de proteínas (figura 1b). Además, desempeñan roles reguladores en la expresión génica al influir en la actividad de otros ARN no codificantes: los microARN (ARNmi) (Zhang et al., 2019) (figura 1b).
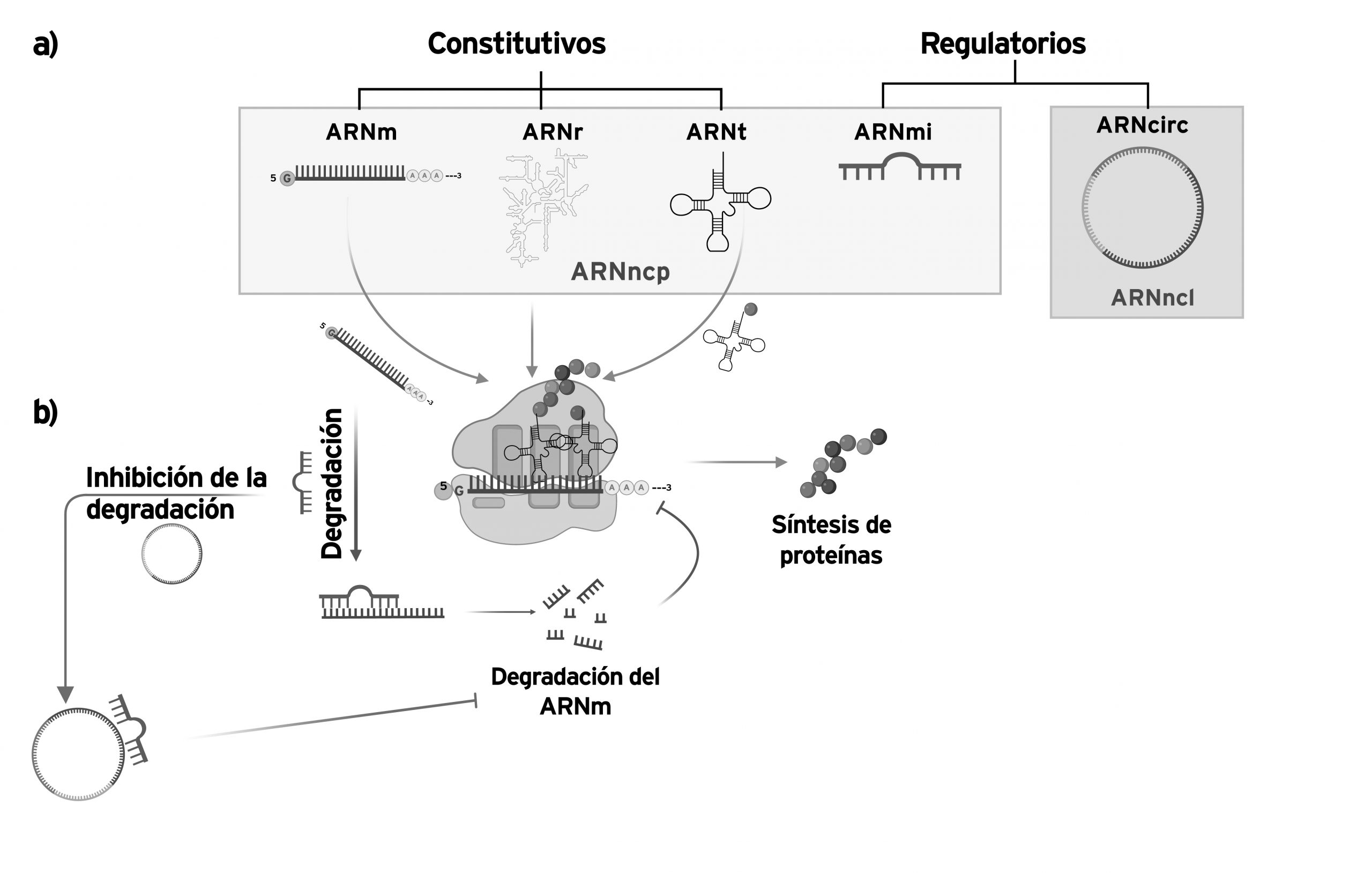
Figura 1. Clasificación de algunos ARN a) Según su función y tamaño: mensajeros (ARNm) y de transferencia (ARNt). Los ARN regulatorios, circulares (ARNcirc) y microARN (ARNmi) se desempeñan en procesos celulares y moleculares. b) La clasificación se basa en la capacidad que tienen los ARN reguladores para afectar el desempeño de los ARN constitutivos a través de su interacción, secuestro y posterior degradación. ARN de transferencia (ARNt) no codificantes pequeños (ARNncp) y largos (ARNncl) (creado con biorender.com).
No obstante, aún existen varios ARN no codificantes que no han sido categorizados o cuyo quehacer no ha sido completamente determinado. Dentro de aquéllos que han despertado interés recientemente se encuentran los circulares (ARNcirc). Éstos han capturado la atención de la comunidad científica debido a su origen en la circularización de fragmentos de ARN y su participación en diversas funciones celulares esenciales. Sin embargo, también intervienen en procesos patológicos, lo que los ha convertido en potenciales marcadores moleculares (biomarcadores) innovadores (Caba et al., 2021; Liu y Chen, 2022).
En este artículo se destacan las características únicas de los ARN circulares, en contraste con otras moléculas de ARN. Se abordan sus actividades biológicas y patológicas, además de explorar su potencial uso e identificación a manera de posibles biomarcadores moleculares.
Aunque los ARNcirc fueron descubiertos hace aproximadamente 30 años en plantas, el interés en ellos ha resurgido debido a que son expresados en una variedad de tejidos eucariotas y altamente conservados entre especies. A pesar de esto, su trabajo biológico sigue siendo enigmático. Son moléculas ubicuas en los mamíferos y su mecanismo de biogénesis es único, aunado a que participan en los procesos de transcripción, traducción, corte, empalme (splicing en inglés) y en la regulación de la expresión génica. Lo relevante es que la cumplen siendo una especie de “esponjas moleculares”, absorbiendo y secuestrando ARNmi, lo que evita que marquen ARNm que han de ser eliminados (J. Li et al., 2020). A la fecha, se han identificado diferentes tipos de ARNcirc, y aunque se ha asociado su presencia con la expresión local de genes y con absorción de otros ARN (esponjas), muchas de sus tareas aún son desconocidas (Li, Yang y Chen, 2018).
BIOGÉNESIS Y ESTRUCTURA DE LOS ARNcirc
Los ARNcirc se distinguen por su estructura circular, a diferencia de otros lineales (figura 1). Antes se pensaba que eran anomalías de un proceso de corte y empalme que forma a los ARNm que participan en la síntesis proteica. Ahora se sabe que se generan mediante un mecanismo alternativo llamado corte y empalme reverso (backsplicing) (Nisar et al., 2021). Su formación comienza cuando la célula necesita producir proteínas y transcribe los genes con el objetivo de obtener precursores de ARNm (preARNm). Éstos se someten a un proceso de empalme, eliminando los fragmentos no codificantes llamados intrones, dando lugar a ARNm maduros que se traducen en proteínas útiles.
El empalme se realiza mediante reacciones químicas entre los extremos 5′ y 3′ del mismo intrón, mediadas por un conjunto de proteínas llamado empalmosoma o espliceosoma. Los ARNcirc se generan a partir de las formas convencionales de ARNm y tienen un impacto en la regulación génica. Su estructura en bucle les proporciona estabilidad y resistencia a la degradación enzimática, a diferencia de los ARNm lineales que son menos estables y más susceptibles a la degradación por ribonucleasas. Esta circularización se considera ventajosa desde una perspectiva evolutiva (Wesselhoeft, Kowalski y Anderson, 2018), pues a diferencia de los ARN lineales tradicionales en su estructura, los circulares forman un bucle cerrado debido a enlaces covalentes especiales entre los extremos. A pesar de esto, la investigación sobre su mecanismo de formación y su versatilidad estructural y funcional sigue en curso mediante estudios experimentales (Nisar et al., 2021).
DESEMPEÑO DE LOS ARNcirc: MÁS ALLÁ DE LO CONVENCIONAL
Aunque la investigación sobre ARNcirc está en curso y sigue siendo un área activa de estudio en la Biología molecular, se han propuesto varias hipótesis sobre por qué las células generan ARNcircs que regulan genes. Algunas de las razones potenciales incluyen:
- Regulación de la expresión génica: los ARNcirc actúan como reguladores de la expresión génica, ya sea inhibiendo o estimulando la traducción de proteínas a partir de ARNm lineales. Algunos ARNcirc funcionarían a modo de “esponjas” de ARNmi o proteínas, atrapándolas y evitando que interactúen con los ARNm, lo que a su vez controlaría la producción de éstas (Zheng et al., 2016).
- Modulación de la actividad de proteínas: algunos ARNcirc podrían interactuar directamente con proteínas específicas y modular sus funciones. Esto afectaría la actividad de éstas y, en última instancia, influiría en diversos procesos celulares (Chen et al., 2018).
- Regulación de la transcripción: se ha descubierto que algunos ARNcirc influyen en la transcripción de genes al interactuar con la maquinaria de transcripción o al modificar la estructura de la cromatina. Esto tendría un impacto en la cantidad de ARNm lineal producido a partir de un gen específico (Li, Yang y Chen, 2018).
- Adaptación a condiciones ambientales cambiantes: los ARNcirc responderían a señales ambientales o condiciones celulares cambiantes. Esto permite a las células ajustar rápidamente su expresión génica en respuesta a estímulos externos o internos, lo que sería crucial en su supervivencia y adaptación (Caba et al., 2021).
ALGUNOS EJEMPLOS DE ARNcirc QUE UNIFORMAN LA EXPRESIÓN GÉNICA
En la actualidad se sabe que los ARNcirc desempeñan un papel importante en la regulación de la expresión génica, y se han propuesto varios mecanismos, como la competencia en el proceso de corte y empalme y su capacidad de hacer de esponjas de ARNmi. Uno de los mecanismos que ha captado mayor atención es su potencial normalización de la actividad de los ARNm y atrapar proteínas y ARNmi relacionados con la transcripción génica. Debido a esta forma de acción se les denomina “esponjas moleculares”, ya que los ARNcirc pueden atrapar múltiples moléculas de ARNmi, lo que temporalmente inhibe ciertas tareas. A pesar de que este mecanismo es complejo y todavía se está comprendiendo en su totalidad, hay muchos detalles que aún están por descubrir (Li et al., 2020).
Los ARNcirc se expresan en una variedad de tejidos, particularmente en el cerebro de diversas especies, incluyendo los seres humanos (Rybak-Wolf et al., 2015), y algunos de ellos se han relacionado con el desempeño de esponja molecular, en patologías del sistema nervioso central (SNC). Un ejemplo es el CiRS-7, que interactúa con el ARNmi llamado miR-7. Esta interacción tiene un impacto en la regulación de vías de señalización relacionadas con la oncogénesis y establece conexiones en el desarrollo neuronal y en males como el Parkinson (Hansen et al., 2013). En el contexto de este padecimiento, la captura de miR-7 por CiRS-7 impide que el ARNmi funcione en sus genes blanco, el caso de snca, el gen que codifica la proteína α-sinucleína, un componente crucial en la progresión de la enfermedad (McMillan et al., 2017). Además, los ARNcirc también desempeñan una labor en la modulación de la maquinaria molecular involucrada en la metilación del ADN (Chen et al., 2018).
Los ARNcirc desempeñan una tarea crucial en la promoción de procesos patológicos, uno de éstos la metástasis en diversos cánceres, al ajustar la maquinaria de metilación y desmetilación del ADN. Un ejemplo es el ARNcirc FECR1, que ha demostrado fomentar la metástasis en cáncer de mama y de pulmón (Li et al., 2019). FECR1 se origina del gen FLI1, el cual también se sobreexpresa en modelos de cáncer de mama, promoviendo la metástasis. El mecanismo de acción de FECR1 implica el ajuste de la expresión de FLI1, previniendo su metilación por la proteína DNMT1 y favoreciendo su expresión al reclutar la proteína TET, que elimina grupos metilo del promotor (Li et al., 2019). Dado el papel de los ARNcirc en diversas patologías, se ha considerado esencial identificar ARNcirc específicos relacionados con estas patologías y así mejorar su diagnóstico y comprensión.
ARNcirc: POTENCIALES MARCADORES DIAGNÓSTICOS EN DIVERSAS ENFERMEDADES Y CONDICIONES PATOLÓGICAS
Los biomarcadores de diagnóstico son elementos únicos que se encuentran en condiciones y tejidos específicos y que resultan fundamentales en los dictámenes médicos. Estos biomarcadores adquieren un valor particular cuando están presentes en muestras fácilmente accesibles, como la sangre (Califf, 2018). Los ARNcirc se encuentran en abundancia en la sangre y otros fluidos corporales (saliva y el líquido cefalorraquídeo), lo que los convierte en candidatos ideales en el cumplimento de este objetivo. Además, su estructura circular les proporciona una mayor estabilidad y resistencia a la degradación. Por esta razón se consideran prometedores biomarcadores en el diagnóstico y pronóstico de diversos malestares (tabla I): cáncer, enfermedades neurodegenerativas, cardiovasculares, infecciosas, metabólicas, autoinmunitarias, entre otras.
En este contexto, los ARNcirc se han convertido en candidatos prometedores en el diagnóstico de enfermedades del SNC. Por ejemplo, en el mal de Parkinson se ha observado una disminución significativa en la expresión de ARNcirc en comparación con individuos sanos (Hanan et al., 2020; Kong et al., 2021). Un ejemplo es el circSLC8A1, que contribuye al estrés oxidativo en el cerebro de los pacientes con Parkinson (Hanan et al., 2020). Sin embargo, la evaluación de circSL-C8A1 en el tejido cerebral tiene limitaciones como biomarcador. Por lo tanto, se han propuesto otros ARNcirc en la sangre periférica, es el caso de circ_0017204 y circ_0004381, capaces de distinguir entre individuos sanos y aquéllos con Parkinson, lo que los convierte en posibles biomarcadores de diagnóstico (tabla I) (Zhong et al., 2021).
Por otro lado, se ha observado una expresión anómala de ARNcirc en muestras de sangre en el cáncer gástrico y pulmonar, lo que sugiere una posible asociación con la agresividad tumoral (Zhang, Yang y Xiao, 2018). Un ejemplo relevante se ha relacionado con el ARNcirc FECR1, propuesto como un biomarcador predictivo y de pronóstico en el cáncer de pulmón, debido a la sobreexpresión de FECR1 en las muestras de sangre periférica de pacientes que lo presentan, en comparación con individuos sanos. Por lo que se ha planteado la hipótesis de que el análisis de los niveles de expresión de FECR1 podría proporcionar información predictiva sobre la expectativa de supervivencia y la respuesta a la quimioterapia (Li et al., 2019).
Los accidentes cerebrovasculares isquémicos y los ataques cardíacos, comunes y con un gran impacto en la expectativa de vida (Mora-Solórzano et al., 2020), generan tejido cicatricial en el corazón, dificultando su función y la recuperación de los pacientes. Los ARNcirc (circHIPK3 y Amotl1) son clave en este proceso. En modelos de ratones con daño cardíaco inducido, la sobreexpresión de Amotl1 reduce la formación de tejido cicatricial y fomenta la proliferación celular (Zeng et al., 2017; Yang et al., 2017), mientras que la de circHIPK3 promueve la cicatrización. Esto muestra cómo ordenar los niveles de ARN circulares puede tener un impacto significativo en el desarrollo y progresión de estas patologías.
Finalmente, es importante destacar que la investigación sobre ARNcirc a modo de biomarcadores está en una fase temprana y sigue evolucionando. Se requiere más investigación que lleve a validar y establecer la utilidad clínica de los ARNcirc en diferentes contextos médicos.
LOS ARNcirc COMO AGENTES TERAPÉUTICOS
La estabilidad inherente de los ARNcirc se ha convertido en una valiosa herramienta en la creación de agentes terapéuticos: vacunas y vehículos de liberación de componentes rehabilitadores (He et al., 2021; Bai et al., 2022). Estos ARNcirc actúan de vectores, entregando secuencias codificantes para proteínas de interés en el interior de las células. Estos vectores están recubiertos con nanopartículas lipídicas que facilitan su internalización en las células, lo que permite la producción de las proteínas que transportan (He et al., 2021) (figura 2a). En este contexto, se están investigando estrategias basadas en ARNcirc como alternativa a las vacunas de ARNm lineales debido a la estabilidad inherente de los ARN circulares. Estas estrategias se han aplicado con éxito en la creación de vacunas contra el SARS-CoV-2, siendo más termoestables y eficaces en la generación de inmunidad. Además, se han obtenido resultados prometedores en estudios preclínicos que ayudan al tratamiento de la leucemia utilizando nanopartículas lipídicas que contienen ARNcirc codificantes para el receptor CD19, lo que ha demostrado ser eficaz en la erradicación del crecimiento de las células cancerosas en estudios con ratones (News y Media, 2020; Garber, 2022).
El uso de ARNcirc como vectores terapéuticos puede ser beneficioso en la entrega de tratamientos y la eliminación de ARNcirc específicos (figura 2b). Por ejemplo, en modelos de cáncer de mama, la sobreexpresión de un ARNcirc llamado Dnmt1circ promueve la proliferación de células cancerosas, pero su inhibición mediante ARNsi la reduce (Du et al., 2018). Aunque prometedores, se requieren ensayos clínicos en humanos que ayuden a evaluar la eficacia y seguridad de estas terapias basadas en ARNcirc.
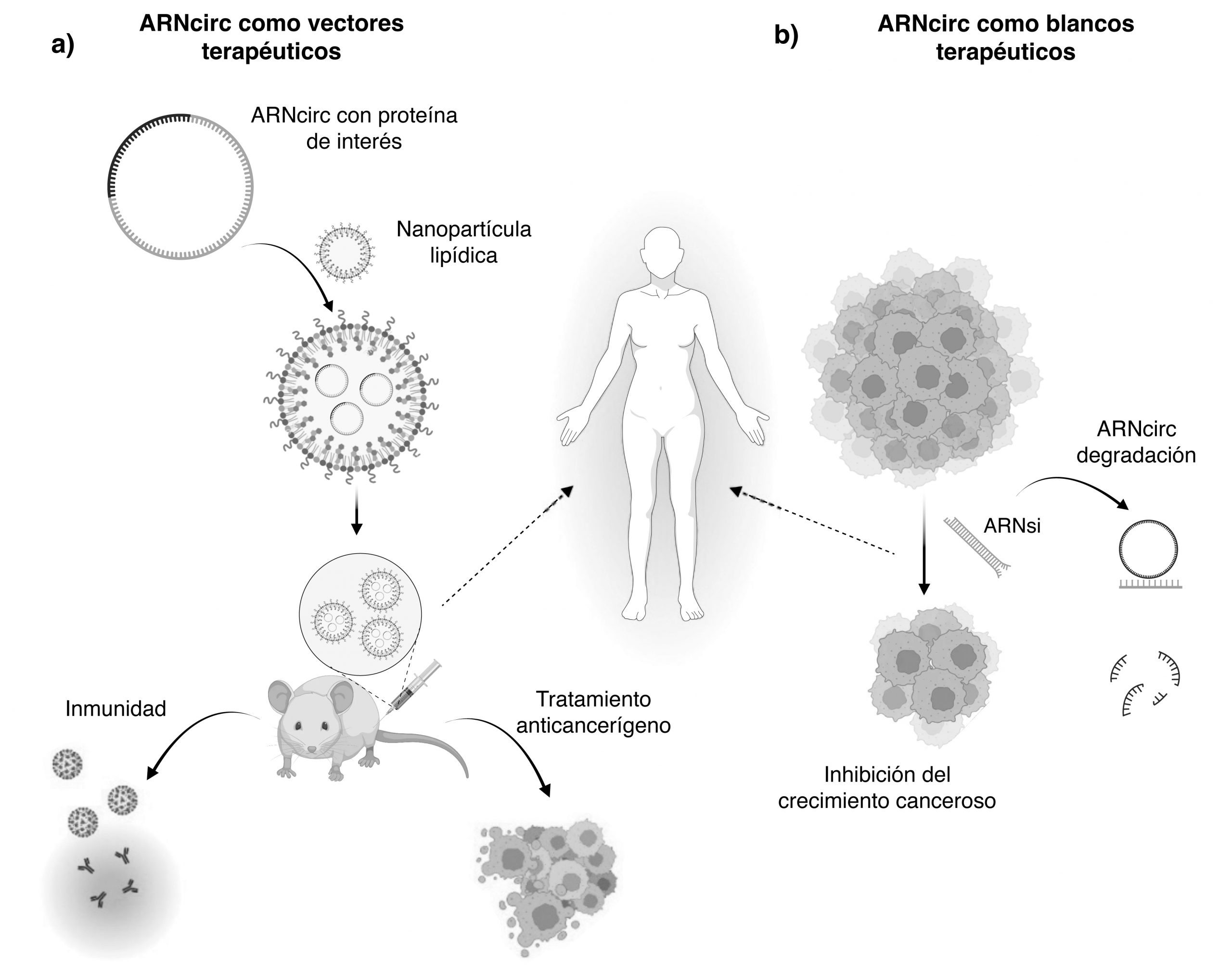
Figura 2. Propuesta terapéuticas de los ARNcirc. a) Los ARNcirc pueden diseñarse de manera que incorporen información genética relacionada con proteínas de efecto terapéutico (antígenos para la creación de vacunas o elementos dirigidos a células cancerosas). b) Otra estrategia implica la eliminación selectiva de ARNcirc específicos, lo cual se lograría mediante su degradación con ARN pequeños de interferencia (ARNsi) (creado con biorender.com).
CONCLUSIONES
El mundo del ARN es amplio y complejo, con actividades que van desde la síntesis de proteínas hasta la regulación celular. Los ARNnc, antes considerados “basura” por su falta de codificación proteica, ahora se reconocen por su papel crucial en ella. Aunque sus orígenes y cometidos son enigmáticos, han ganado importancia en enfermedades, como los ARNcirc que podrían ser biomarcadores útiles. Además, se anticipa que las terapias basadas en ARN, especialmente ARNcirc, aprovecharán su estabilidad estructural para la expresión estable de elementos terapéuticos que modulen funciones celulares, mejorando la calidad de vida y enriqueciendo nuestras herramientas contra diversas afecciones.
* Universidad Autónoma Metropolitana, Cuajimalpa de Morelos, Ciudad de México. Contacto: csamano@cua.uam.mx
REFERENCIAS
Bai, Y., Liu, D., He, Q., et al. (2022). Research progress on circular RNA vaccines, Frontiers in Immunology, 13, 1091797.
Caba, L., Florea, L., Gug, C., et al. (2021). Circular RNA-Is the Circle Perfect?, Biomolecules, Multidisciplinary Digital Publishing Institute, 11(12), 1755.
Califf, R.M. (2018). Biomarker definitions and their applications, Experimental Biology and Medicine , 243(3), 213-221.
Chen, N., Zhao, G., Yan, X., et al. (2018). A novel FLI1 exonic circular RNA promotes metastasis in breast cancer by coordinately regulating TET1 and DNMT1, Genome Biology, 19(1), 218.
Dai, X., Zhang, S. y Zaleta-Rivera, K. (2020). RNA: interactions drive functionalities, Molecular Biology Reports, 47(2), 1413-1434.
Du, W.W., Yang, W., Li, X., et al. (2018). A circular RNA circ-DNMT1 enhances breast cancer progression by activating autophagy, Oncogene, 37(44), 5829-5842.
Ezkurdia, I., Juan, D., Rodríguez, J.M., Frankish, A., et al. (2014). Multiple evidence strands suggest that there may be as few as 19,000 human protein-coding genes, Human Molecular Genetics, 23(22), 5866-5878.
Garber, K. (2022). Orna Therapeutics: circular logic, Nature Biotechnology, https://doi. org/10.1038/d41587-022-00005-1
Hanan, M., Simchovitz, A., Yayon, N., et al. (2020). A Parkinson’s disease CircRNAs Resource reveals a link between circSLC8A1 and oxidative stress, EMBO Molecular Medicine, 12(11), e13551.
Hansen, T.B., Jensen, T.I., Clausen, B.H., et al. (2013). Natural RNA circles function as efficient microRNA sponges, Nature, 495(7441), 384-388.
He, A.T., Liu, J., Li, F. y Yang, B.B. (2021). Targeting circular RNAs as a therapeutic approach: current strategies and challenges, Signal Transduction and Targeted Therapy, 6(1), 185.
Hombach, S. y Kretz, M. (2016). Non-coding RNAs: Classification, Biology and Functioning, Advances in Experimental Medicine and Biology, 937, 3-17.
Human. (s/f). Statistics about the current GENCODE Release (version 45), https:// www.gencodegenes.org/human/stats.html
Kong, F., Lv, Z., Wang, L., et al. (2021). RNA-sequencing of peripheral blood circular RNAs in Parkinson disease, Medicine, 100(23), e25888.
Li, J., Sun, D., Pu, W., et al. (2020). Circular RNAs in Cancer: Biogenesis, Function, and Clinical Significance, Trends in Cancer Research, 6(4), 319-336.
Li, L., Li, W., Chen, N., et al. (2019). FLI1 Exonic Circular RNAs as a Novel Oncogenic Driver to Promote Tumor Metastasis in Small Cell Lung Cancer, Clinical Cancer Research: An Official Journal of the American Association for Cancer Research, 25(4), 1302-1317.
Liu, C.-X., y Chen, L.-L. (2022). Circular RNAs: Characterization, cellular roles, and applications, Cell, 185(13), 2390.
Li, X., Yang, L. y Chen, L.-L. (2018). The Biogenesis, Functions, and Challenges of Circular RNAs, Molecular Cell, 71(3), 428-442.
Luo, Y.-H., Yang, Y.-P., Chien, C.-S., et al. (2020). Plasma Level of Circular RNA hsa_ circ_0000190 Correlates with Tumor Progression and Poor Treatment Response in Advanced Lung Cancers, Cancers, 12 (7), https://doi.org/10.3390/cancers12071740.
Mora-Solórzano, L., Gutiérrez-Díaz, G.I., Gudiño-Amezcua, D.A., et al. (2020). Características clínicas de pacientes con infarto agudo de miocardio tratados con trombólisis en urgencias, Revista Médica del Instituto Mexicano del Seguro Social, Publicidad Permanyer, SLU, 58(2), 100-107.
News & Media. (2020). Orna Therapeutics, 18 diciembre, https://www.ornatx.com/ news-and-media/?pag=1&category=21
Nisar, S., Bhat, A.A., Singh, M., et al. (2021). Insights Into the Role of CircRNAs: Biogenesis, Characterization, Functional, and Clinical Impact in Human Malignancies, Frontiers in Cell and Developmental Biology, 9, 617281.
Nurk, S., Koren, S., Rhie, A., et al. (2022). The complete sequence of a human genome, Science, 376(6588), 44-53.
Panni, S., Lovering, R.C., Porras, P., et al. (2020). Non-coding RNA regulatory networks, Biochimica et Biophysica Acta, Gene Regulatory Mechanisms, 1863(6), 194417.
Rybak-Wolf, A., Stottmeister, C., Glažar, P., et al. (2015). Circular RNAs in the Mammalian Brain Are Highly Abundant, Conserved, and Dynamically Expressed, Molecular Cell, 58(5), 870-885.
Saito, H. (2022). The RNA world ‘hypothesis’, Nature Reviews. Molecular Cell Biology, 23(9), 582.
Sang, Q., Liu, X., Wang, L., et al. (2018). CircSNCA downregulation by pramipexole treatment mediates cell apoptosis and autophagy in Parkinson’s disease by targeting miR-7, Aging, 10(6), 1281-1293.
Statello, L., Guo, C.-J., Chen, L.-L., et al. (2021). Author Correction: Gene regulation by long non-coding RNAs and its biological functions, Nature Reviews. Molecular Cell Biology, 22(2), 159.
Wesselhoeft, R.A., Kowalski, P.S., y Anderson, D.G. (2018). Engineering circular RNA for potent and stable translation in eukaryotic cells, Nature Communications, 9(1), 2629.
Yang, Z.-G., Awan, F.M., Du, W.W., et al. (2017). The Circular RNA Interacts with STAT3, Increasing Its Nuclear Translocation and Wound Repair by Modulating Dnmt3a and miR-17 Function, Molecular Therapy: The Journal of the American Society of Gene Therapy, 25(9), 2062-2074.
Zeng, Y., Du, W.W., Wu, Y., et al. (2017), A Circular RNA Binds To and Activates AKT Phosphorylation and Nuclear Localization Reducing Apoptosis and Enhancing Cardiac Repair, Theranostics, 7(16), 3842- 3855.
Zhang, P., Wu, W., Chen, Q., et al. (2019). Non-Coding RNAs and their Integrated Networks, Journal of Integrative Bioinformatics, 16(3), https://doi.org/10.1515/jib-2019-0027
Zhang, Z., Yang, T., y Xiao, J. (2018). Circular RNAs: Promising Biomarkers for Human Diseases, EBioMedicine, 34, 267-274.
Zheng, Q., Bao, C., Guo, W., et al. (2016). Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs, Nature Communications, 7, 11215.
Zhong, L., Ju, K., Chen, A., et al. (2021). Circulating CircRNAs Panel Acts as a Biomarker for the Early Diagnosis and Severity of Parkinson’s Disease, Frontiers in Aging Neuroscience, 13, 684289.
Recibido: 05/12/2022.
Aceptado: 14/09/2023.