Se requieren dos para bailar tango: interacción de dos aminoácidos de Antennapedia con TFIIEβ para el desarrollo de patas en Drosophila
Claudia Altamirano-Torres* ORCID 0000-0001-5919-0660
Carolina Hernández-Bautista* ORCID 0000-0001-8576-4592
Diana Reséndez-Pérez* ORCID 0000-0002-4709-7677
CIENCIA UANL / AÑO 26, No.120, julio-agosto 2023
DOI: https://doi.org/10.29105/cienciauanl26.120-5
RESUMEN
Las homeoproteínas dirigen el desarrollo embrionario de los organismos mediante la unión del homeodominio al DNA y su interacción con otras proteínas. Antennapedia (Antp) se encarga de formar patas y alas en el tórax e interactúa con TFIIEβ a través del homeodominio en D. melanogaster. En este trabajo mostramos que la interacción de únicamente dos aminoácidos de Antp con TFIIEβ son necesarios para la formación de patas. La función de estas interacciones contribuye al complejo y fascinante rompecabezas de la especificidad de los genes Hox en el control genético del desarrollo en Drosophila.
Palabras clave: desarrollo embrionario, Drosophila, homeoproteínas, regulación génica, interacción.
ABSTRACT
Homeoproteins direct embryo development of organisms through homeodomain DNA-binding to DNA and protein-protein interactions. Antennapedia (Antp) directs leg and wing formation in the thorax and interacts with TFIIEβ via the homeodomain in D. melanogaster. Here we found that the interaction of two aminoacids of Antp with TFIIEβ are necessary for leg formation. The function of these interactions contributes to the complex and fascinating puzzle of Hox gene specificity in the genetic control Drosophila development.
Keywords: embryonic development, Drosophila, homeoproteins, genetic regulation, interaction.
El desarrollo de los organismos inicia con una sola célula totipotente denominada cigoto, la cual rápidamente se segmenta en dos, cuatro, ocho, etcétera, para dar lugar a las células diferenciadas que constituirán los tejidos y órganos, culminando en un organismo completo. Las células formadas durante la segmentación se organizan y adquieren identidades específicas de acuerdo con su posición a lo largo de los distintos ejes corporales, es decir, establecen todos los tipos celulares que van a constituir los órganos y tejidos desde la cabeza hasta la parte más posterior del cuerpo; este proceso embrionario es conocido como morfogénesis (Gilbert y Barresi, 2016).
Una de las grandes interrogantes en biología del desarrollo es cómo las células totipotenciales pueden convertirse en las miles de células diferenciadas requeridas en tiempo y forma precisos para integrar el cuerpo de un organismo. Para responder esta pregunta es necesario el uso de modelos experimentales que nos permitan diseccionar los pasos que, a nivel molecular, se requieren para la adquisición de destinos celulares y morfogénesis. La pequeña mosca de la fruta (Drosophila melanogaster) es un excelente modelo del desarrollo por su fácil manejo en el laboratorio y rápido ciclo de vida, pero principalmente porque estamos evolutivamente relacionados: los mismos genes que especifican su desarrollo también especifican el de los mamíferos, incluidos los humanos.
Un grupo de genes de particular importancia en el desarrollo son los genes homeóticos, que fueron identificados por Walter Gehring y por Edward B. Lewis, este último galardonado con el Premio Nobel de Fisiología y Medicina en 1995. En Drosophila, estos genes codifican a las homeoproteínas que se encargan de especificar la identidad corporal a lo largo de un embrión, es decir, se encargan de que la cabeza se ubique en su lugar y tenga ojos, antenas y probóscide, o que en el tórax se formen patas y alas (Gehring, 1987). Se les considera reguladores maestros del desarrollo por su papel fundamental en la morfogénesis, controlando funciones celulares diversas como migración, morfología celular, proliferación, apoptosis y diferenciación (Cerdá-Esteban y Spagnoli, 2014). Estos genes no sólo se encuentran en la mosca, sino también en otros vertebrados como el humano, e inclusive microorganismos eucariotas como levaduras (Heffer y Pick, 2013).
Las homeoproteínas deben su nombre a que contienen un homeodominio (HD) de 60 aminoácidos que sirve como un sitio de unión al DNA y que les confiere función como factores transcripcionales, prendiendo o apagando genes específicos para construir patas, alas o cualquier otra estructura corporal, y especificar la posición en la que dichas estructuras deben ubicarse (Gehring et al., 1995). Los HD están altamente conservados, es decir, su secuencia aminoacídica es casi idéntica entre diferentes homeoproteínas y diferentes organismos, además, todas reconocen la misma secuencia en el DNA, planteando la denominada “paradoja Hox”: cómo proteínas tan similares son capaces de especificar de manera precisa identidades corporales tan diversas como la cabeza o las extremidades. Entre los modelos propuestos para explicar la especificidad funcional se encuentra la interacción de estas proteínas con otros factores de regulación transcripcional, estableciendo redes a través de una amplia variedad de interacciones proteína-proteína denominadas en conjunto interactoma Hox (Bobbola et al., 2017).
Drosophila posee ocho homeoproteínas que especifican las características morfológicas de la cabeza, los tres segmentos torácicos y los ocho segmentos abdominales que conforman el cuerpo de la mosca. Específicamente, la homeoproteína Antennapedia (Antp) se encarga de dar identidad al segundo segmento torácico, donde promueve el desarrollo del segundo par de patas y las alas de la mosca (Cásares y Mann, 1998). Se sabe que Antp interacciona con diferentes factores transcripcionales, y de particular interés ha sido su interacción con miembros de la maquinaria basal como BIP2 o TFIIEβ (Prince et al., 2008; Baeza et al., 2015). Recientemente, mediante ensayos de complementación bimolecular fluorescente (BiFC), determinamos que Antp interactúa con TFIIEβ a través de los residuos 32 y 36 en la hélice 2 del HD en cultivo celular e in vivo (Altamirano-Torres et al., 2018). La maquinaria basal es un grupo de factores transcripcionales que se encarga de reclutar la RNA polimerasa II para iniciar la transcripción de cualquier gen codificante y se encuentran y funcionan en todos los tejidos. Por el contrario, cada homeoproteína se encuentra en un tejido específico en el que inducirá la formación de estructuras corporales particulares.
La relación de las homeoproteínas con la maquinaria basal dada por interacciones proteína-proteína ha sido de gran importancia, ya que la regulación transcripcional ocurre principalmente sobre el ensamblaje de la maquinaria basal, y el estudio de estas interacciones nos permite ampliar el conocimiento sobre los mecanismos de especificidad funcional de las homeoproteínas. En nuestro laboratorio analizamos la interfase de interacción entre Antp y TFIIEβ para determinar su importancia funcional durante el desarrollo de Drosophila, y encontramos que sólo dos aminoácidos de Antp son necesarios para que ocurra esta interacción y permita la transformación antena-pata.
DOS AMINOÁCIDOS ESPECÍFICOS DE ANTP SON NECESARIOS PARA LA INTERACCIÓN CON TFIIEβ Y LA FORMACIÓN DE PATAS
Previamente, en el laboratorio analizamos la interacción proteína-proteína de Antp con TFIIEβ mediante mutaciones en Antp para definir la región responsable de esta interacción (Altamirano-Torres et al., 2018), y encontramos que el HD es la región de Antp que interacciona con TFIIEβ. Como la estructura del HD consiste en tres hélices α(H1,H2yH3), y debido a que la H3 es responsable de la unión al DNA, se analizó el papel de las hélices 1 y 2 en esta interacción. Los resultados obtenidos mostraron que la interacción depende únicamente de dos aminoácidos en la hélice 2 del HD. Estos resultados fueron validados usando la doble mutante Antp![]() , en la que se sustituyó la isoleucina 32 y la histidina 36 de la hélice 2 por alaninas. El cambio por alanina se seleccionó debido a su carácter neutro y a su predicción para estructurarse en hélices α, lo que permitiría mantener la estructura del HD y neutralizar químicamente las posiciones 32 y 36 de la hélice 2. De esta manera, con la doble mutante, logramos establecer que la falta de los residuos 32 y 36 no permite la interacción de Antp con TFIIEβ usando BiFC (figura 1A) (Altamirano-Torres et al., 2018).
, en la que se sustituyó la isoleucina 32 y la histidina 36 de la hélice 2 por alaninas. El cambio por alanina se seleccionó debido a su carácter neutro y a su predicción para estructurarse en hélices α, lo que permitiría mantener la estructura del HD y neutralizar químicamente las posiciones 32 y 36 de la hélice 2. De esta manera, con la doble mutante, logramos establecer que la falta de los residuos 32 y 36 no permite la interacción de Antp con TFIIEβ usando BiFC (figura 1A) (Altamirano-Torres et al., 2018).
Para responder la pregunta de ¿qué función puede tener esta interacción en el desarrollo de la mosca y qué importancia tienen esos dos aminoácidos en la hélice 2 de Antp?, realizamos el análisis mediante la generación de moscas transgénicas y cruzas genéticas. Con las cruzas podemos dirigir las proteínas de interés a cualquier tejido de la mosca y observar el efecto analizando el fenotipo producido. La presencia de las homeoproteínas en tejidos donde normalmente no se encuentran puede cambiar la identidad de manera dramática, cambiando un segmento por otro, lo que se conoce como transformaciones homeóticas.
Normalmente, Antp se encarga de formar patas en el tórax, pero cuando dirigimos esta proteína a las antenas, las transforma en patas, generando una mosca ¡con patas en la cabeza! (figura 1B). La transformación de antenas en patas es una función homeótica de Antp que utilizamos en el laboratorio para analizar su función ectópica y, además, definir la importancia de regiones proteicas, o como en este caso, la de los residuos 32 y 36 de la hélice 2 en la interacción Antp-TFIIEβ y en la transformación antena-pata.
Con el uso de cruzas genéticas expresamos Antp o la mutante doble (Antp![]() ) junto con TFIIEβ de forma ectópica, específicamente en las antenas de la mosca. La interacción con TFIIEβ causó una completa transformación de la antena, con estructuras características de una pata de insecto como fémur, tarso y uña (figura 1B). Sorprendentemente, cuando dirigimos a TFIIEβ y la doble mutante Antp
) junto con TFIIEβ de forma ectópica, específicamente en las antenas de la mosca. La interacción con TFIIEβ causó una completa transformación de la antena, con estructuras características de una pata de insecto como fémur, tarso y uña (figura 1B). Sorprendentemente, cuando dirigimos a TFIIEβ y la doble mutante Antp![]() a la antena no ocurre la transformación antena-pata (figura 1C). Es importante mencionar que la expresión de TFIIEβ por sí sola no tuvo ningún efecto en la transformación de la antena (Altamirano-Torres et al., 2018), por lo que estos resultados claramente muestran que la interacción de Antp con TFIIEβ es requerida para su función en la formación de patas ectópicas en la antena, y que la mutación de únicamente dos aminoácidos son clave para el correcto funcionamiento de Antp en Drosophila.
a la antena no ocurre la transformación antena-pata (figura 1C). Es importante mencionar que la expresión de TFIIEβ por sí sola no tuvo ningún efecto en la transformación de la antena (Altamirano-Torres et al., 2018), por lo que estos resultados claramente muestran que la interacción de Antp con TFIIEβ es requerida para su función en la formación de patas ectópicas en la antena, y que la mutación de únicamente dos aminoácidos son clave para el correcto funcionamiento de Antp en Drosophila.
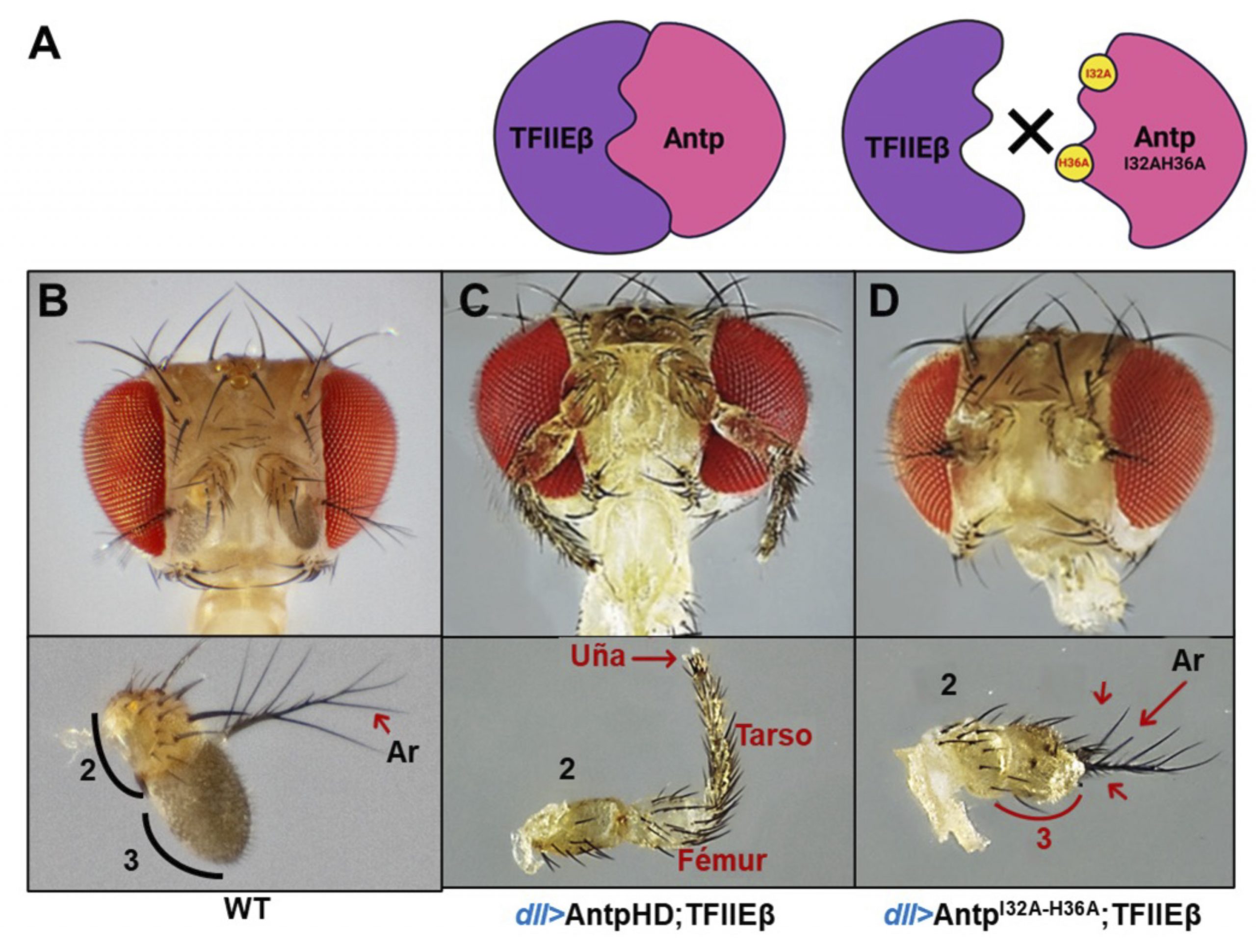
Figura 1. La interacción de Antp con TFIIEβ es necesaria para la transformación de antenas en patas en D. melanogaster. Las proteínas de interacción AntpHD y TFIIEβ se expresaron en la antena mediante cruzas genéticas. A) Antp y TFIIEβ interactúan a través de dos aminoácidos en la hélice 2 del HD de Antp. B) Antena normal (WT) de Drosophila. C) La interacción AntpHD-TFIIEβ indujo la transformación homeótica completa de la antena en una pata. D) La coexpresión de la doble mutante AntpI32A-H36A y TFIIEβ disminuyó drásticamente la transformación homeótica de la antena, con sólo un leve engrosamiento del tercer segmento. Los segmentos transformados se indican mediante líneas y leyendas en rojo (2, segundo segmento de la antena; 3, tercer segmento de la antena; Ar, arista).
Los resultados obtenidos concuerdan con los de otros laboratorios que han encontrado interacciones entre otros HD también con TFIIEβ, o con otros factores transcripcionales como Med19 (Boube et al., 2014). Dado que los residuos 32 y 36 en la hélice 2 de Antp están altamente conservados, y físicamente expuestos en la cara opuesta a la unión al DNA, éstos mismos podrían estar involucrados en la interacción de otras homeoproteínas con TFIIEβ. Tales interacciones muestran de forma muy clara la función dual del HD para interactuar otras proteínas a través de la hélice 2, y unirse al DNA mediante la hélice 3.
Nuestros resultados sugieren que la interacción de Antp con TFIIEβ contribuye a la regulación transcripcional que permite la actividad funcional de Antp, y surge la pregunta de cómo se lleva a cabo dicha regulación en conjunto con la maquinaria basal de transcripción. Sabemos que Antp se encarga de la formación de patas apagando genes de antena como Hth y Dll (Emerald et al., 2003), así que la interacción de Antp con TFIIEβ podría estar cooperando para el apagado de los genes de antena y el encendido de genes de pata (figura 2). Aunque es necesario llevar a cabo más experimentos para definir con claridad el mecanismo molecular de acción de esta interacción durante la transcripción génica, nuestros resultados sientan las bases para estudiar a la hélice 2 del HD como una región clave de interacción con otras proteínas.
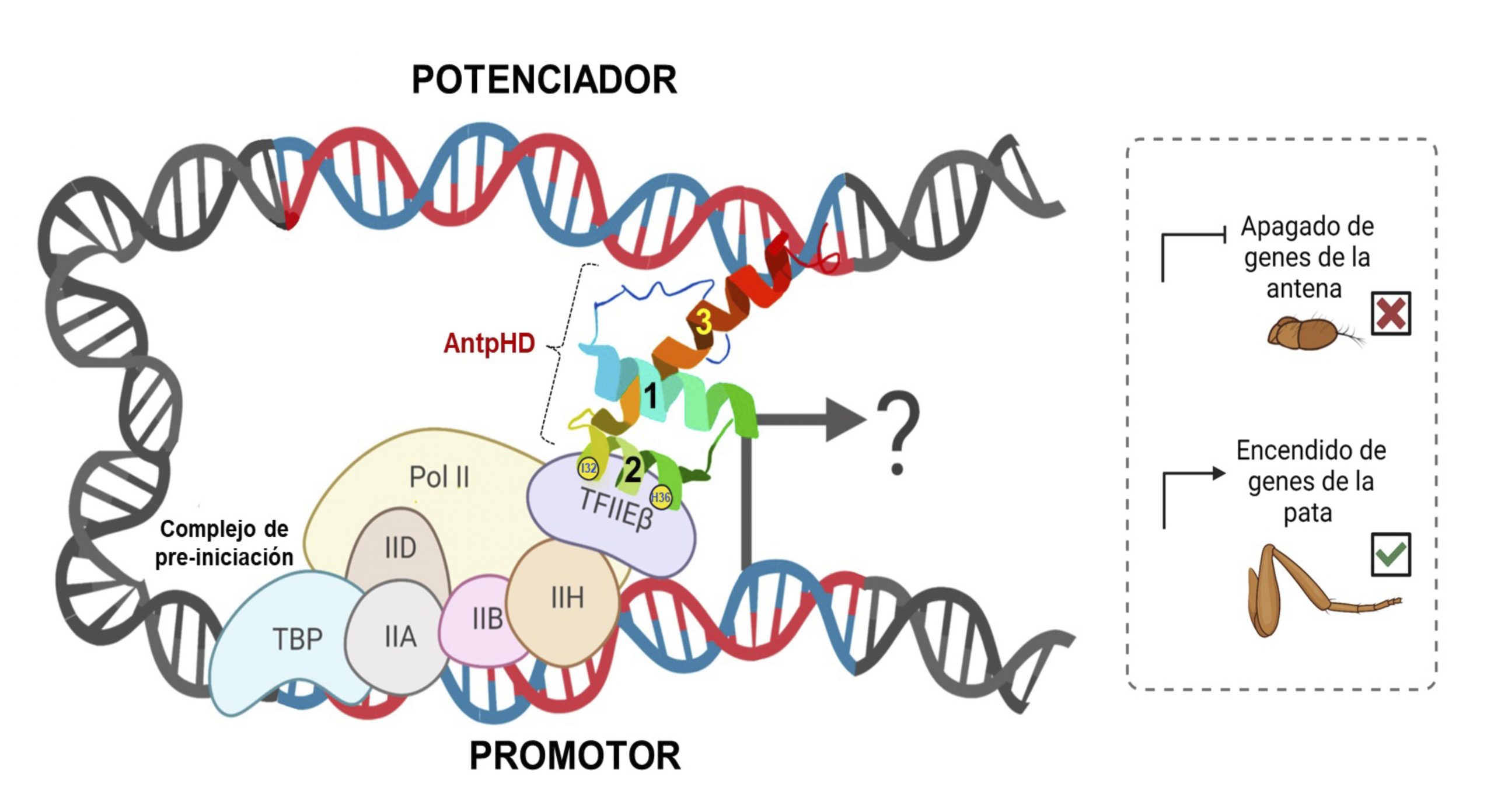
Figura 2. La interacción de Antp con TFIIEβ a través de los aas 32 y 36 de la hélice 2 regula transcripción génica. La hélice 2 del HD de Antp interactúa con TFIIEβ en la maquinaria basal de transcripción. Tal interacción podría, en primer lugar, servir como puente entre el promotor y el potenciador “enhancer” tejido-específico a los que Antp se une. Una vez dada, la interacción podría ser responsable de apagar a los genes de antena y encender los de pata para generar la formación de los apéndices en la mosca.
CONCLUSIONES
En este trabajo determinamos que la interacción Antp-TFI- IEβ es necesaria para la actividad funcional de Antp en la formación ectópica de patas en Drosophila, y que esta función depende únicamente de dos aminoácidos (32 y 36) en la hélice 2 del HD de Antp. En conjunto, nuestros resultados contribuyen al complejo y fascinante rompecabezas de la especificidad de los genes Hox en la ejecución del control genético del desarrollo en Drosophila. Dada la alta conservación de las homeoproteínas, el estudio de estas interacciones y su función en Drosophila pueden ser extrapolados al desarrollo embrionario de otros organismos, incluido el humano.
* Universidad Autónoma de Nuevo León, San Nicolás de los Garza, México.
Contacto: diana.resendezpr@uanl.edu.mx
REFERENCIAS
Altamirano-Torres, C., Salinas-Hernández, J.E., Cárdenas-Chávez, D.L., et al. (2018). Transcription factor TFIIEβ interacts with two exposed positions in helix 2 of the Antennapedia homeodomain to control homeotic function in Drosophila. Plos one. 13(10):e0205905.
Baëza, M., et al. (2015). Inhibitory activities of short linear motifs underlie Hox interactome specificity in vivo. Elife. 4:e06034. https://elifesciences.org/articles/06034
Bobbola, N., y Merabet, S. (2017). Homeodomain proteins in action: similar DNA binding preferences, highly variable connectivity. Curr Op in Genetics & Dev. 43:1-8. https://www.sciencedirect.com/science/article/pii/S0959437X16301265
Boube, M., et al. (2014). Drosophila melanogaster Hox transcription factors access the RNApolymerase II machinery through direct homeodomain binding to a conserved motif of mediator subunit Med19. PLoS Genetics. 10(5):e1004303. https://journals. plos.org/plosgenetics/article?id=10.1371/journal.pgen.1004303
Brand, A.H., y Perrimon, N. (1993). Targeted expression as a means of altering cell fates and generating dominant phenotypes. Development.118:401-415. https://doi.org/10.1242/dev.118.2.401
Casares, F., y Mann. RS. (1998). Control of antennal versus leg development in Drosophila. Nature. 392:723-726. https://www.nature.com/articles/33706
Cerdá-Esteban, N., y Spagnoli, F.M. (2014). Glimpse into Hox and tale regulation of cell differentiation and reprogramming. Developmental Dynamics. 243(1):76-87. https://doi.org/10.1002/dvdy.24075
Emerald, B.S., et al. (2003). Distal antenna and distal antenna related encode nuclear proteins containing pipsqueack motifs involved in antenna development in Drosophila. Development. 130:1171-1180. https://doi.org/10.1242/dev.00323
Gehring, W.J. (1987). Homeoboxes in the study of development. Science. 236:1245-1252. https://science.sciencemag.org/content/236/4806/1245.long
Gehring, W.J., et al. (1995). Homeodomain-DNA recognition. Cell. 78:211-223. https://doi. org/10.1016/0092-8674(94)90292-5
Gilbert, S.F., y Barresi, M.J.F. (2016). Developmental Biology, 11th Edit. Sinnauer Associates Inc.
Lewis, E.B. (1978). A gene complex controlling segmentation in Drosophila. Nature, 276(5688):565- 570. https://www.nature.com/articles/276565a0
Prince, F., et al. (2008). The YPWM motif links Antennapedia to the basal transcriptional machinery. Development. 135:1669-1679. https://doi.org/10.1242/dev.018028
