Proteoma y vías de señalización inducidas por AAS en células que expresan el VHC
ADRIANA SÁNCHEZ GARCÍA*, HERMINIA G. MARTÍNEZ RODRÍGUEZ*, CLARA P. RÍOS IBARRA*,
ANA R. RINCÓN SÁNCHEZ**, ANA M. RIVAS ESTILLA*
CIENCIA UANL / AÑO 18, No. 71, ENERO-FEBRERO 2015
La infección crónica por el virus de la hepatitis C (VHC) puede desencadenar enfermedades hepáticas graves como cirrosis y carcinoma hepatocelular; afecta a 3% de la población mundial y se considera un problema grave de salud pública. (1-4)
El VHC pertenece a la familia Flaviviridae, con genoma de RNA de cadena sencilla y polaridad positiva. El tratamiento estándar, hasta 2011, consistía en la combinación de interferón α pegilado y ribavirina. Recientemente se ha sugerido el uso de inhibidores de la proteasa viral (boceprevir o telaprevir); sin embargo, no en todos los pacientes tratados se consigue una respuesta virológica sostenida. (5)
En la actualidad, los modelos de replicón genómico y subgenómico han hecho posible el desarrollo de agentes que bloquean específicamente la replicación viral. (6,7) A este respecto, Trujillo et al., con un sistema de replicón subgenómico tratado con ácido acetilsalicílico (AAS), reportaron la disminución del RNA y proteínas del VHC; (8) sin embargo, el mecanismo implicado está pobremente definido. Mediante el análisis exhaustivo de perfiles proteicos de expresión diferencial entre muestras normales, patológicas o expuestas a un tratamiento obtenidos con electroforesis bidimensional (2-DE) o chips de proteínas, la proteómica provee información de biomarcadores y blancos asociados a enfermedades. (9,10)
Con base en lo anterior, examinamos la expresión diferencial de proteínas de la línea celular Huh7, que expresa las proteínas no estructurales del VHC (Huh7 replicón-VHC) y la línea Huh7 parental expuestas a AAS, a fin de elucidar el mecanismo relacionado con la disminución de la expresión del VHC. Observamos que la presencia de AAS induce un patrón diferencial de proteínas en células tratadas, y promueve la activación de proteínas relacionadas con progresión celular, proliferación, inhibición de la apoptosis y crecimiento, al mismo tiempo que disminuye la expresión del VHC.
OBJETIVO
Identificar perfiles diferenciales de proteína en células Huh7, que expresan proteínas no estructurales del VHC y en células Huh7 parental expuestas a AAS, para elucidar el mecanismo involucrado en la disminución de la replicación del VHC mediada por AAS.
MATERIAL Y MÉTODOS
Cultivo celular y tratamiento con AAS 4 mM
Se utilizó la línea celular de hepatocarcinoma Huh7, que expresa las proteínas no estructurales del VHC genotipo 1b (replicón subgenómico) y la línea celular Huh7 parental (sin replicón). Las células se mantuvieron en medio ADMEM (GIBCO-BRL, suplementado con 2% de suero bovino fetal (GIBCO-BRL), 1% de aminoácidos no esenciales, 100 U de penicilina G y 100 μg/mL de estreptomicina por mL a 37°C y 5% de CO2. Para el tratamiento, las células se sembraron un día antes y se trataron con 4 mM de AAS (Sigma-Aldrich)(0-72 h).
Extracción de proteína total
Las células se trataron con 4 mM de AAS y se incubaron por 0 a 72 h. A cada tiempo, las células se cosecharon y se extrajeron las proteínas totales con el kit MicroRotoforTM Lysis (Bio-Rad), seguido de la purificación de proteínas con el estuche ReadyPrep 2D Cleanup (Bio-Rad). La cuantificación se realizó mediante Bradford.
Electroforesis bidimensional y análisis proteómico
Para la electroforesis 2D, 120 μg de proteína se aplicaron en tiras IPG (Bio-Rad) de 17 cm pH 3-10. La separación en primera dimensión se hizo en el equipo Protean IEF Cell (Bio-Rad), con un gradiente de 10,000 V en 2.5h y 40,000 V/h. Las tiras IPG se equilibraron después en buffer (urea 6 M, SDS 2%, Tris-HCl 50 mM, pH 8.8, glicerol 30%), con DTT a 0.5% y buffer con iodoacetamida a 4.5%. La separación en segunda dimensión se realizó con el sistema Protean II XI Cell (Bio-Rad), en poliacrilamida a 12%, 150 V y 4°C. Se adicionó un marcador de peso molecular (PM) y punto isoeléctrico (pI) de siete proteínas conocidas, para establecer puntos de referencia en el análisis subsecuente. Todos los geles se prepararon por triplicado.
Análisis proteómico e identificación de las proteínas sobreexpresadas
Las proteínas se revelaron con tinción de nitrato de plata (Silver Stain Plus Kit, Bio-Rad). Los perfiles de proteínas obtenidos en geles se digitalizaron en el densitómetro GS-800 (Bio-Rad), y el análisis bidimensional se hizo con el software PDQuest V8.0.1 (Bio-Rad). Se identificaron diferencias en las proteínas expresadas de acuerdo con Gasteiger et al., (11) en las que una herramienta bioinformática de proteínas calcula el PM y pI estimado de un dato específico de la base de datos Swis-Prot/TrEMBL, o una secuencia específica de aminoácidos.
Brevemente, las proteínas fueron identificadas y elucidadas bioinformáticamente por su pI y PM con el programa TagIdent (Uniprot Consortium), (http:/ /www.expasy. org/tools/pi_tools.html). TagIdent realiza la identificación de proteínas al comparar etiquetas de secuencia contra proteínas de la base de datos Swiss-Prot de una o más especies de organismos dentro de un intervalo de pI y PM. Además, se realizó un análisis de componente principal (ACP) para determinar las proteínas de mayor relevancia en las condiciones probadas para determinar los coeficientes factoriales (nivel de correlación y componente principal) con el programa Unscrambler 9.8. Los datos reportados son los promedios ± desviación estándar de tres experimentos independientes. El análisis estadístico incluyó la prueba de t de Student (p<0.05).
RESULTADOS
Colectamos el proteoma celular después de cada periodo de incubación (0-72h), con el tratamiento de AAS 4 mM, enfocándonos en la máxima inhibición de VHC en las células Huh7 replicón (48 y 72h postratamiento) (*p<0.05). (8) Los patrones bidimensionales diferenciales mostraron entre 674 y 1416 proteínas por gel, con la mayoría localizada en la región de alto PM y de pI ligeramente ácido, distribuidas en un rango de 10 a 140 kDa (figura 1A y 1B). El mismo estándar interno se usó en todas las muestras. El promedio del número de manchas que representan proteínas individuales o proteínas con migración similar obtenidas fue de 923 y 816 para células Huh7 parental y Huh7 replicón, respectivamente. Luego de comparar y normalizar a condiciones basales las manchas de proteína, se identificaron numerosas manchas expresadas diferencialmente (sub y sobreexpresadas) entre ambas líneas celulares tratadas con AAS.
Detectamos 37 manchas de proteína diferencialmente expresadas de manera cualitativa, y 343 manchas de proteína expresadas diferencialmente de manera cuantitativa. La comparación de los geles de ambas líneas celulares (parental y replicón) mostró 24 manchas de proteína sobreexpresadas significativamente en las células replicón (3 veces, t-test, p<0.05).
En la figura 1 se muestran perfiles de expresión proteica representativos de ambas líneas celulares en ausencia y presencia de AAS 4 mM a diferentes tiempos de exposición. Encontramos proteínas diferencialmente expresadas, tanto cualitativa como cuantitativamente a 24, 48 y 72 h. A 24 y 48 h, se observa el mayor número de proteínas diferencialmente expresadas con significancia estadística (tabla I). Los resultados se evaluaron mediante ACP para distinguir proteínas correlacionadas en el contexto del modelo de estudio, y reducir y discriminar entre el gran número de variables aquéllas con mayor trascendencia. La varianza explicada (EV = 1/n •XtX) con dos componentes principales (CP1 y CP2) fue la siguiente: a las 24h observamos una EV de 86 y 12%; a las 48 h la EV fue de 57 y 32%, y a las 72h fue de 64 y 20% para las células tratadas o no con AAS, respectivamente.
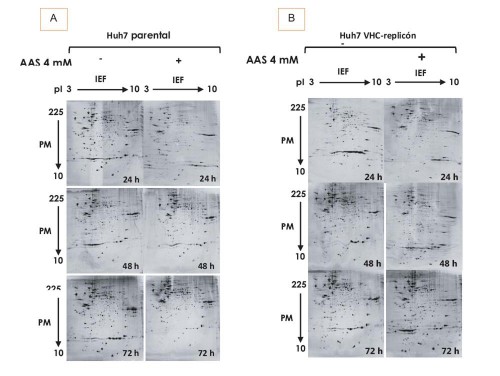
Fig. 1. Separación bidimensional de proteínas de células Huh7 parental y Huh7 replicón-VHC obtenida luego de la 2-DE. Las proteínas se separaron en tiras IPG con gradiente de pH lineal 3-10, con 120 μg de proteína, seguida de SDS-PAGE a 12% (20 cm x 20 cm 1.5 mm). Los geles se tiñeron con nitrato de plata (Bio-Rad). A) Análisis bidimensional del extracto de proteína de células Huh7 parental tratadas con AAS 4 mM a diferentes tiempos. B) Análisis bidimensional de la proteína extraída de células Huh7 replicón-VHC tratadas con AAS 4 mM a diferentes tiempos.
En ambos tipos celulares, se analizaron las diferencias en la expresión proteica en respuesta al tratamiento en las 1416 proteínas detectadas con una prueba de t independiente. Con interés, observamos que las células parentales tuvieron una respuesta diferente a la exposición a AAS, puesto que el total y la suma de proteínas diferencialmente expresadas a los tres tiempos fue mayor que las proteínas diferencialmente expresadas en las células replicón (167 vs. 57 proteínas, respectivamente) (tabla I). Por otra parte, las células replicón tienen menos proteínas diferencialmente expresadas a 72h postratamiento (tabla I).

Tabla I. Proteínas diferencialmente expresadas encontradas en la línea celular Huh7 parental y replicón luego del tratamiento con AAS. DIF* Proteínas diferencialmente expresadas. En paréntesis se muestra el número de proteínas estadísticamente significativas con t-test entre los grupos con y sin AAS al mismo tiempo de evaluación. AAS= 4mM
El análisis proteómico reportó 289 proteínas diferencialmente expresadas entre las células replicón-VHC, en presencia o ausencia de AAS. Con respecto a la identificación de proteínas involucradas en disminución mediada por AAS de la replicación del VHC-1b, encontramos que la mayoría de las proteínas sobreexpresadas a las 24h del tratamiento con AAS se relaciona con proliferación celular, mostrando la expresión de las proteínas MTMR6, FAM22, HDGF y HCF-1. Luego de 48 h de exposición a AAS, observamos la sobreexpresión de angiostatina, PI4KA y STAT-1 y, finalmente, a las 72h postratamiento se observó sobreexpresión de adenilsuccinato sintasa (proteína relacionada con la síntesis de purinas en hígado), 2’,3’-di-deoxiadenosina, ubiquitin-protein-ligasa E6A, adenilsuccinato liasa y nibrina (tabla II).
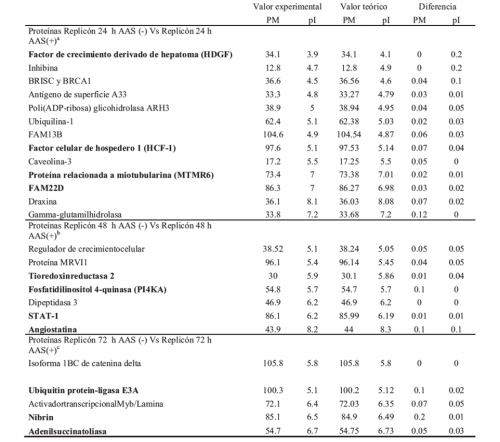
Tabla II. Proteínas diferencialmente expresadas encontradas en la línea celular Huh7 parental y replicón luego del tratamiento con AAS. DIF* Proteínas diferencialmente expresadas. En paréntesis se muestra el número de proteínas estadísticamente sigificativas con t-test entre los grupos con y sin AAS al mismo tiempo de evaluación. AAS= 4mM.
DISCUSIÓN
A pesar de los extensos estudios de los antiinflamatorios no establecidos (AINEs), poco se sabe de sus efectos en las infecciones virales. Nuestro grupo demostró previamente que el efecto antiviral de AAS es parcialmente dependiente de la inhibición de la actividad de cicleoxigenasa-2 (COX-2) y la síntesis de prostaglandinas en el sistema celular de replicón subgenómico del VHC 1b.8 Con base en el hallazgo de Trujillo et al., en este trabajo abordamos la búsqueda de efectores que pudieran intervenir en el resultado observado para obtener más información del mecanismo antiviral, aplicando técnicas proteómicas y herramientas bioinformáticas para analizar globalmente el perfil de expresión proteica de la línea celular humana de hepatocarcinoma Huh7, en presencia y ausencia de tratamiento con AAS. Varios de nuestros hallazgos concordaron con datosreportados en que se empleó un sistema de cultivo celular con el replicón subgenómico/genómico del VHC. (12-14) Estas diferencias de expresión entre las condiciones estudiadas coinciden con reportes previos en la expresión diferencial de las células Huh7 y las células que expresan las proteínas no estructurales del VHC; (15) sin embargo, adicionalmente, se encontraron diferentes patrones de proteína en los hepatocitos que expresaban las proteínas del VHC y células tratadas con AAS.
La infección por el VHC afecta el ciclo celular y distintas vías de señalización, y contribuye a la patogénesis viral. Con base en lo anterior, se podrían explicar los resultados que obtuvimos al comparar las proteínas diferencialmente expresadas de las células Huh7 parental y Huh7 replicón, en respuesta a la exposición de AAS (tabla I). El tratamiento con AAS afecta la expresión proteica de la célula hospedera; pero, al mismo tiempo, el perfil de proteínas es afectado por la presencia de las proteínas virales. Se encontró que el VHC promueve la activación de proteínas relacionadas en la proliferación y progresión del ciclo celular. MTMR6, HDGF y HCF- 1 se han asociado previamente con la inhibición de la apoptosis, la unión a oncoproteínas, el estímulo del crecimiento celular y la progresión del ciclo celular, respectivamente. Luego de 48 h postratamiento con AAS, el virus continúa promoviendo la proliferación y la progresión de la célula huésped por inducción de la proteína PI4Ka y angiostatina. Sin embargo, de manera simultánea, se identificaron proteínas relacionadas con el arresto celular y apoptosis como STAT1 y tioredoxin-reductasa. Previamente, Hartman et al. reportaron que STAT1 en presencia de interferon (IFN) promueve el arresto del ciclo celular y la apoptosis. (16) Por su parte, Hinze et al. y Ganther et al. sugieren que la tioredoxin-reductasa participa como antioxidante, al reducir el proceso viral. (17,18) Nuestros resultados muestran que a las 72h del tratamiento con AAS, se sobreexpresan proteínas como la ubiquitin-protein-ligasa E6A, adenilsuccinato liasa y nibrina que podrían participar en la inhibición de la replicación del VHC. A este respecto, Shirakura et al. mencionan que la ubiquitinprotein- ligasa E6A participa en la degradación de la proteína de la cápside del virus. (19) La adenil-succinato liasa se requiere para la activación de la vía antiviral de la proteína 2’,3’di-dideoxiadenosina. Otra proteína sobreexpresada fue la nibrina, de la cual se ha reportado que posee actividad oncogénica y un rol en la reparación de las roturas de doble hebra.
Las proteínas identificadas podrían sugerir que en las horas tempranas, luego de la infección, el virus promueve la desregulación de la proliferación y el ciclo celular en su beneficio y que, después del tratamiento con AAS, la célula infectada comienza el proceso de defensa. Deben realizarse más experimentos, a fin de validar tanto los hallazgos del análisis proteómico como del análisis in silico para seguir esclareciendo el panorama molecular de la infección por VHC.
CONCLUSIÓN
La caracterización parcial de proteínas diferencialmente expresadas, durante el tratamiento con AAS, apunta hacia la identificación de candidatos relacionados a su actividad antiviral. El análisis proteómico efectuado provee información global de cómo se modifica el proteoma de las células Huh7 replicón, en respuesta a la exposición con AAS, así como indicios que contribuyen al entendimiento del mecanismo de replicación y patogénesis del VHC.
RESUMEN
El AAS disminuye la expresión del VHC por mecanismos aún desconocidos. Se compararon perfiles de expresión proteómica en células de hepatocarcinoma (Huh7) y células que expresan proteínas no estructuradas del VHC (Huh7-VHC) tratadas con AAS 4mM. Los perfiles obtenidos mediante electroforesis bidimensional fueron analizados por pI y PM. Se identificaron diferentes patrones entre células Huh7- VHC tratadas y no tratadas con AAS. Entre las proteínas diferencialmente expresadas encontramos proteínas relacionadas con proliferación celular (MTMR6, FAM22, HDGF y HCF-1) y sobreexpresión de angiotensina, PI4KA y STAT-1. A las 72h, identificamos sobreexpresión de adenil-succinato sintasa, 2’-3’-di-deoxiadenosina, proteína ligasa de ubiquitina E6A, adenilsuccinato-liasa y nibrina. El AAS induce diferentes patrones proteicos en células Huh7- VHC, promoviendo la activación de proteínas relacionadas con progresión celular, reparación de DNA, inhibición de apoptosis y estimulación del crecimiento.
Palabras clave: Ácido acetilsalicílico (AAS), Virus de la hepatitis C (VHC), Proteoma, Proteómica, Electroforesis bidimensional (2-DE).
ABSTRACT
ASA has been shown to downregulate HCV expression; however, the involved mechanisms are unknown. We used proteomic analysis to compare protein expression profiles between human hepatocarcinome cells (Huh7) and Huh7-HCV cells harboring expression of non-structural HCV proteins to elucidate the mechanisms involved in ASAmediated downregulation of HCV replication. Cell lines were treated with 4 mM ASA and harvested to isolate total proteins, which were resolved by 2-DE. Gels were analyzed and proteins elucidated by pI and MW patterns. Different protein patterns among hepatocytes expressing HCV-proteins in ASA treated and untreated cells were found. Among proteins differentially expressed we found proteins related to cellular proliferation (MTMR6, FAM22, HDGF y HCF-1) and overexpression of angiotensin (PI4KA y STAT-1). We found that ASA induces different protein patterns in Huh7-HCV cells promoting activation of proteins involved in cell progression, repair of double strand breaks, proliferation, inhibition of apoptosis and growth stimulation at the same time that it decreased HCV expression.
Key words: Acetylsalicylic acid (ASA), Hepatitis C virus (HCV), Proteome, Proteomics, Bidimensional electrophoresis (2-DE).
AGRADECIMIENTOS
Los autores agradecen al Programa de Apoyo a la Investigación Científica y Tecnológica (Paicyt) SA1010-04 y Promep 103.5/04/2590, por el apoyo económico brindado.
————————-
El presente artículo está basado en la investigación “Análisis Proteómico e identificación de proteínas y vías de señalización inducidas por ácido acetilsalicílico en células que espresan el virus de la hepatitis C”, galardonado con el Premio de Investigación UANL 2014 en la categoría de Ciencias de la Salud, otorgado en sesión solemne del Consejo Universitario de la UANL, en agosto de 2014.
* Universidad Autónoma de Nuevo León, Facultad de Medicina.
** Universidad de Guadalajara, Guadalajara.
Contacto: amrivas@yahoo.ca
REFERENCIAS
1. Alter M. Epidemiology of hepatitis C virus infection. World J Gastroenterol 2007; 13: 2436-41.
2. Drazan K. Molecular biology of hepatitis C infection. Liver Transplantation 2000; 6: 396-406.
3. Sabahi A. Hepatitis C virus entry: the early stops in the viral replication cycle. Virology Journal 2009; 6: 117.
4. Sy T., Jamal M. Epidemiology of Hepatitis C Virus (HCV) Infection. Int J Med Sci 2006; 3: 41-6.
5. Chevaliez S., Pawlotsky J. Hepatitis C virus: Virology, diagnosis and management of antiviral therapy. World J Gastroenterol 2007; 13: 2461-6.
6. Blight K., Kolykhalov A., Rice C. Efficient initiation of HCV RNA replication in cell culture. Science 2000; 290: 1972-4.
7. Wakita T., Pietschmann T., Kato T., Date T., Miyamoto M., Zhao Z., Murthy K. et al. Production of infectious hepatitis C virus in tissue culture from a cloned viral genome. Nat Med 2005; 11: 791-6.
8. Trujillo K.C., Rincón A.R., Martínez H.G., Bosques F.J., Ramos J., Barrera H., Rojkind M., Rivas A.M. Acetylsalicylic acid inhibits hepatitis C virus RNA and proteína expresión through cyclooxygenase-2 signaling pathways. Hepatology 2008; 47: 1462-72.
9. Molloy M., Witzmann F. Proteomics: Technologies and applications. Briefings in functional genomics and proteomics 2002; 1: 23-9.
10. Motonari T., Yasuhiro K., Yuuichiro Y., Norio I., Toshifusa T., Isao S., Kiwamu O. et al. Proteomic profiling of heat shock protein 70 family members as biomarkers for hepatitis C virus-related hepatocellular carcinoma. Proteomics 2003; 3: 2487-93.
11. Gasteiger E., Hoogland C., Gattiker A., Duvaud S., Wilkins M.R., Appel R.D., Bairoch A. Protein identification and analysis tools on the ExPASy Server. In: Walker JM (ed). NJ: Humana Press Inc., 2005, p 571-607.
12. Xun M., Zhao S., Cao C. et al. Proteomic analysis of Huh-7 cells harboring in Vitro-transcribed full-length hepatitis C virus 1b RNA. Acta Pharmacologica Sinica 2008; 29: 720-7.
13. Diamond D., Syder A., Jacobs J., Sorensen C., Walters K., Proll S., McDermott J., Gritsenko M., Zhang Q., Zhoao R., Metz T., Camp D., Waters K., Smith R., Rice C., Katze M. Temporal proteome and lipidote profiles reveals Hepatitis C Virus-associated reprogramming of hepatocellular metabolism and bioenergetics. PLoS Pathogens 2010; 6: 1-18.
14. Joyce M.A., Walters K.A., Lamb S.E., Yeh M.M., Zhu L.F., Kneteman N., Doyle J.S. et al. HCV induces oxidative and ER stress and sensitizes infected cells to apoptosis in SCID/Alb-uPA mice. PLoS Pathog 2009; 5: e1000291.
15. Fang C., Yi Z., Liu F., Lan S., Wang J., Lu H., Yang P., Yuan Z. Proteome analysis of human liver carcinoma Huh7 cells harboring hepatitis C virus subgenomic replicon. Proteomics 2006; 6: 519-27.
16. Hartman S.E., Bertone P., Nath A.K., Royce T.E., Gerstein M., Weissman S., Snyder M. Global changes in STAT target selection and transcription regulation upon interferon treatments. Genes Dev 2005; 19: 2953-68.
17. Hinze K., Wald K., Zeng H., Jeffery E., Finley J. Thiredoxin reductase in human hepatoma cells is transcriptionally regulated by sulforaphane and other electrophiles via an antioxidant response element. J Nutr 2003; 133: 2721-7.
18. Ganther H.E. Selenium metabolism, selenoproteins and mechanisms of cancer prevention: complexitites with thioredoxin reductase. Carcinogenesis 1999; 20: 1657-66.
19. Shirakura M., Murakami K., Ichimura T., Suzuki R., Shimoji T., Fukuda K., Abe K. et al. E6AP ubiquitin ligase mediates ubiquitylation and degradation of hepatitis C virus core protein. J Virol 2007; 81: 1174-85.
Recibido: 18/07/14
Aceptado 18/08/14