Secuencia de la tanasa de Aspergillus niger GH1 y producción de la enzima en Pichia pastoris
José Antonio Fuentes Garibay*, Martha Guerrero Olazarán*, José María Viader Salvadó*
CIENCIA UANL / AÑO 20, No. 83, ENERO-MARZO 2017
Resumen
En este trabajo se describen las secuencias nucleotídicas y aminoacídicas y la probable estructura molecular de la tanasa del hongo xerófito Aspergillus niger GH1. Además, se construyeron cepas de la levadura Pichia pastoris para producir de forma extracelular la enzima, la cual se caracterizó bioquímicamente y sus propiedades se compararon con las de la tanasa nativa. La tanasa producida en este trabajo podría ser utilizada en el procesado de alimentos y bebidas a bajas temperaturas.
Palabras Clave: tanasa, Aspergillus niger GH1, mode-laje molecular, Pichia pastoris.
Abstract
In this paper, we describe the gene and amino acid sequences, and the possible molecular structure of the tannase of the Aspergillus niger GH1. In addition, we engineered Pichia pastoris strains with a synthetic gene to produce and secrete the enzyme, Furthermore, the produced recombinant tannase was biochemically characterized, and its properties were compared to those of the native tannase from A. niger GH1. The tannase produced in this study could be used in the processing of food and beverages at low temperatures.
Key words: tannase, Aspergillus niger GH1, molecular modelling, Pichia pastoris.
Las tanin acil hidrolasas o tanasas (EC 3.1.1.20) son enzimas que catalizan la hidrólisis de los enlaces éster en galotaninos, taninos complejos y ésteres de ácido gálico, produciendo ácido gálico como producto principal. Estas enzimas son usadas en la industria de alimentos durante la fabricación del té, clarificación del vino y jugo de fruta, y para la reducción de efectos antinutricionales de los taninos en nutrición animal (Aguilar y Gutiérrez, 2001). Además, el ácido gálico se utiliza para la síntesis del antioxidante propilgalato y el antibiótico trimetroprim. A pesar de las aplicaciones de las tanasas, el uso práctico de estas enzimas es limitado debido a la falta de un procedimiento económico para su producción a gran escala.
Recientemente, Aspergillus niger GH1, un hongo xerófilo, ha sido reportado como un productor de tanasa. Este microorganismo fue aislado del semidesierto mexicano y tolera condiciones extremas de la región (45-15ºC) (Cruz et al., 2005). La tanasa de A. niger GH1 ha demostrado ser muy efectiva para el tratamiento de desechos ricos en taninos, potenciando la actividad biológica de té y la producción de importantes y potentes compuestos fenólicos antioxidantes. En comparación con otras tanasas, la de A. niger GH1 mostró alta estabilidad a pH, temperatura y otros compuestos que generalmente se encuentran en los sistemas ricos en taninos. Basados en estas propiedades, esta enzima puede ser especialmente utilizada en aplicaciones industriales.
Por otro lado, la levadura metilotrófica Pichia pastoris se utiliza frecuentemente como un hospedero para la producción de proteínas recombinantes extracelula-res, ya que puede crecer en medios definidos simples y alcanzar altas densidades celulares, produciendo de esta manera altos niveles de proteínas extracelulares recombinantes (Sreekrishna, 2010).
En este trabajo se describen por primera vez las secuencias nucleotídicas y aminoacídicas, y la probable estructura molecular de la tanasa de A. niger GH1. Además, se construyeron cepas de P. pastoris para producir de forma extracelular la enzima, la cual se caracterizó bioquímicamente y sus propiedades se compararon con las de la tanasa nativa.
Materiales y Métodos
Para determinar la secuencia nucleotídica de la tanasa de A. niger GH1, se sintetizó dicha secuencia por PCR utilizando DNA genómico de A. niger GH1 y oligonucleótidos dirigidos hacia el inicio y al final de la secuencia que codifica para la proteína madura. El producto amplificado se ligó en el vector pGEM y se transformaron células de Escherichia coli. Cinco plásmidos prove-nientes de diferentes colonias de E. coli se secuenciaron en el Instituto de Fisiología Celular (UNAM), usando los oligonucleótidos T7 y SP6. Cuatro de estos plásmidos se secuenciaron nuevamente empleando oligonucleótidos internos diseñados con base en la secuencia nucleotídica obtenida con los oligonucleótidos T7 y SP6. Se determinó la secuencia nucleotídica consenso a partir de las 18 secuencias nucleotídicas, empleando el módulo CAP (Contig Assembly Program) del programa BioEdit v7.0.8.0. Esta secuencia y la secuencia aminoacídica deducida se compararon con las secuencias reportadas en la base de datos GenBank mediante las herramientas BLAST del NCBI y se determinaron los porcentajes de identidad con otras tanasas fúgicas mediante el programa Clustal Omega. Se hizo un análisis de dominios funcionales de la tanasa de A. niger GH1 por comparación de la secuencia aminoacídica obtenida con la base de datos de familias de proteínas Pfam. Se construyó un modelo molecular de la proteína con el servidor Phyre (2), y con el programa Swiss-PdbViewer/DeepView 4.1 se analizaron aspectos estructurales y funcionales de la tanasa de A. niger GH1. Con el ser-vidor NetNGlyc 1.0 se predijeron los posibles sitios de N-glicosilación.
Para la construcción de cepas recombinantes de P. pastoris KM71 productoras de la tanasa de A. niger GH1, se diseñó un gen artificial (ANTgs) que codifica para la tanasa de A. niger GH1 mediante el uso de co-dones preferenciales de P. pastoris y la inclusión de la secuencia prepro del factor alfa de Sacharomyces cerevisiae. El gen artificial fue sintetizado, clonado en el vector pUC57 y subclonado en pPIC9 para obtener el plásmido pPIC9ANTgs. Se linearizó el plásmido pPI-C9ANTgs construido con la enzima SalI y se transformó la cepa KM71 de P. pastoris mediante electropo-ración. De las cepas construidas se caracterizaron su genotipo (aox1 y ANT+) mediante PCR y su fenotipo mediante la evaluación de la expresión de la secuencia clonada en cultivos a nivel matraz, a través de la actividad enzimática extracelular de tanasa.
Con el fin de realizar una caracterización bioquímica de la enzima producida, con una preparación enzimática obtenida por ultrafiltración del medio de cultivo libre de células de los cultivos en matraz, se determinó la presencia de posibles N-glicosilaciones evaluando el cambio en la migración en geles de poliacrilamida de muestras tratadas y sin tratar con la glucosidasa endo Hf. Además, se determinó la presencia de una posible estructura bicatenaria de la tanasa producida. Se evaluó el efecto del pH y la temperatura en la actividad enzimática de tanasa, la estabilidad de la enzima a cuatro y 30°C, la actividad específica y los parámetros cinéticos Km y Vmax.
Resultados
Determinación de la secuencia nucleotídica y aminoacídica de la tanasa nativa de A. niger GH1
La secuencia nucleotídica de la tanasa de A. niger GH1 madura tuvo una longitud 1,686 pb, sin intrones, y codifica para una proteína de 562 aminoácidos. Esta secuencia nucleotídica se registró en la base de datos GenBank con la clave de acceso KP273835.
La secuencia de aminoácidos de la tanasa de A. niger GH1 mostró identidades de 78.6-99.4% con respecto a las seis tanasas fúngicas más similares: Aspergillus kawachii IFO 4308 (GenBank GAA91900.1, 99.4%), A. niger CBS 513.88 (GenBank XP_001402486.1, 98.0%), A. niger ATCC 1015 (GenBank EHA22262.1, 97.1%), Aspergillus terreus NIH2624 (GenBank XP_001216558.1, 80.0%), A. niger (GenBank ABX89592.1, 79.2%) y la tanasa y feruloil esterasa de Aspergillus ruber CBS 135680 (GenBank EYE96818.1, 78.6%); con identidades en las secuencia nucleotídicas que van en un intervalo de 73.3 a 98.6% para las mismas tanasas fúngicas. El análisis con Pfam mostró que la secuencia de la tanasa de A. niger GH1 tiene un dominio del residuo 53 al 545 que pertenece a la familia Tanasa PF07519 en donde se agrupan otras tanasas, feruloil estearasas y varias proteínas de bacterias de función desconocida. Esta familia es un miembro del clan AB_hidrolasa (CL0028) cuyo dominio catalítico se encuentra en una amplia gama de enzimas, ya que este clan contiene 67 miembros. En la familia PF07519 se encuentran descritas 15 arquitecturas de organización de dominios, presentando la tanasa de A. niger GH1 la arquitectura más sencilla de un único dominio.
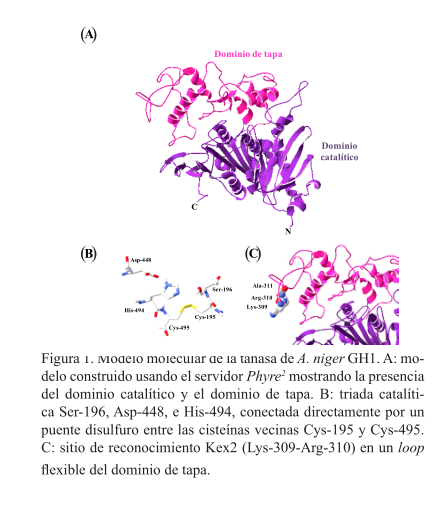
El programa Phyre2 realizó un modelo molecular con todos los aminoácidos de la tanasa de A. niger GH1, 512 residuos (91%) fueron modelados con base en la estructura 3WMT, que pertenece a una feruloil esterasa (Suzuki et al., 2014), y 50 residuos (9%) fue-ron modelados mediante técnicas de ab initio. El modelo molecular de la tanasa de A. niger GH1 (figura 1A) mostró la presencia de 16 hélices alfa y 16 hojas beta que abarcan 29 y 13% de la proteína, respectiva-mente; que forman dos dominios estructurales, uno de ellos con un plegamiento tipo α/β-hidrolasa que constituye el dominio catalítico, y el otro un dominio tipo tapa que cubre el sitio catalítico. A partir del modelo molecular y mediante comparación con la estructura 3WMT, se proponen los residuos Ser-196, Asp-448, e His-494 como los que constituyen la triada catalítica. Además, estos residuos de serina e histidina pudieran estar directamente conectados mediante un puente di-sulfuro entre las cisteínas que se encuentran contiguas a la serina e histidina, Cys-195 y Cys-495 (figura 1B), formando el motivo estructural descrito recientemente (CS-D-HC) (Suzuki et al., 2014). El modelo molecular también mostró que uno de los sitios de reconocimiento tipo Kex2 (Lys-309-Arg-310, figura 1C) se encuentra en un loop flexible del dominio de tapa.
Construcción de cepas recombinantes de P. pastoris portadoras del fragmento ANTgs (KM71ANT)
La secuenciación del DNA del plásmido pUC57 confirmó la secuencia nucleotídica correcta del gen sintético ANTgs. El gen sintético de la tanasa junto con la secuencia nucleotídica que codifica para la péptido prepro del factor alfa de S. cerevisiae tuvo un tamaño de 1,961 pb. El análisis mediante PCR del plásmido recombinante, usando los oligonucleótidos de AOX, mostró un producto de 2,158 pb que confirmó la construcción del plásmido pPIC9ANTgs. La transformación de células de P. pastoris KM71 con el plásmido pPIC9ANTgs previamente digerido con SalI generó 15 colonias transformadas His+. El análisis mediante PCR con los oligonucleótidos de AOX mostró una banda de 2,158 pb que corresponde a la secuencia que codifica para la secuencia prepro del factor alfa (255 pb), a la secuencia del gen sintético que codifica para la tanasa de A. niger GH1 (1,689 pb) y los fragmentos del sitio múltiple de clonación del vector pPIC9 (21 pb), promotor de AOX1 (94 pb) y del terminador de la transcripción (99 pb).
Producción y caracterización bioquímica de la tanasa de A. niger GH1 recombinante
Después de 48 h de cultivo de una cepa KM71ANT en condiciones de inducción del gen heterólogo, el medio de cultivo libre de células mostró una actividad volumétrica de tanasa de 0.57 U/ml y una concentración de proteínas de 46.71 mg/L. Estos resultados corroboraron la capacidad de la cepa KM71ANT de producir y secretar al medio de cultivo la tanasa de A. niger GH1 recombinante, y por lo tanto confirmaron su fenotipo ANT+. Con el proceso de ultrafiltración que se aplicó al medio de cultivo libre de células se obtuvo un concentrado de tanasa (25.7 U/ml) que se empleó para la caracterización bioquímica de la tanasa recombinante producida.
En el análisis de SDS-PAGE (figura 2), la tanasa recombinante sin tratamiento con la enzima endo Hf mostró dos bandas anchas de masas moleculares aparentes de 45.0-54.8 y 41.8-45.0 kDa, con una intensidad mayor en las masas moleculares de 48.7 y 42.4 kDa (figura 2, carril 2). Después de la N-desglicosilación con endo Hf, las dos bandas se movieron a posiciones con masas moleculares aparentes de 34.6 y 30.3 kDa, respectiva-mente (figura 2, carril 3).
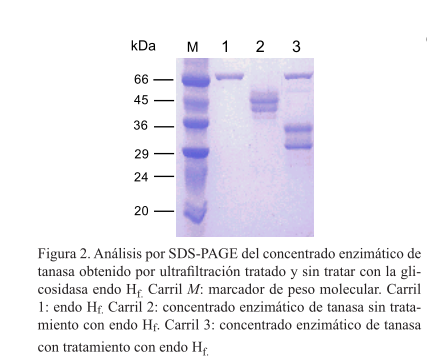
Estos resultados indican claramente que la tanasa está altamente N-glicosilada y que posee dos cadenas polipeptídicas. Además, el análisis de la secuencia aminoacídica de la tanasa recombinante con el programa NetNGlyc 1.0 mostró que la tanasa madura de A. niger GH1 tiene 11 sitios potenciales de N-glicosilación: 19NGTL, 54NVTV, 130NGSI, 237NATI, 265NLTS, 280NYTS, 304NGSV, 383NVTY, 451NTTY, 508NATV y 535NSSF.
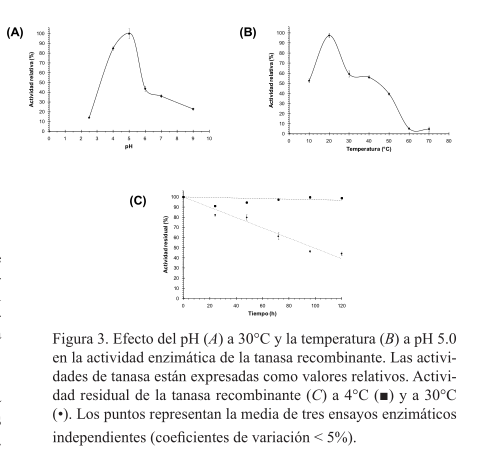
La tanasa recombinante producida tuvo un pH óptimo de 5.0 (figura 3A) y mostró más de 80% de su máxima actividad en el intervalo de pH entre 4.0 y 5.0. Además, mostró actividad en un amplio intervalo de temperaturas (10-50°C) con más de 40% de su máxima actividad y una temperatura óptima de 20°C (figura 3B). La actividad residual (figura 3C) en los ensayos de estabilidad siguieron cinéticas de orden cero con una velocidad de reacción de 0.01 y 0.48%/h a 4 y 30ºC, respectivamente, disminuyendo a 98.9 y 44.0% después de 120 h.
Un cromatograma de intercambio aniónico del con-centrado de la tanasa recombinante mostró dos picos predominantes mediante detección por UV a 280 nm. La actividad específica (20ºC, pH 5.0) en el máximo de uno de estos picos fue de 50 U/mg de proteína. La actividad de la tanasa recombinante siguió una cinética típica de Michaelis-Menten a 20 y 30°C (r2= 0.947 y 0.978, respectivamente). Las constantes de Michaelis (Km) y la velocidad máxima de reacción (Vmax) fueron estimadas en valores de 1.98 ± 0.50 mM y 2.01 ± 0.14 µmol/min, y 0.18 ± 0.04 mM y 0.48 ± 0.02 µmol/min a 20 y 30°C, respectivamente.
Discusión
Recientemente, el hongo xerófilo A. niger GH1 ha sido descrito como un productor de tanasa (Cruz et al., 2005). Las propiedades bioquímicas de la tanasa nativa de A. niger GH1 también han sido descritas (Renovato et al., 2011). En el presente trabajo se reportan las secuencias nucleotídica y aminoacídica de la tanasa madura de A. niger GH1. Además, se utilizó el siste-ma de expresión de P. pastoris para producir la enzima, utilizando un gen sintético, y se caracterizó la tanasa recombinante producida.
Las secuencias nucleotídica y aminoacídica de la ta-nasa de A. niger GH1 resultaron ser similares, pero no idénticas, a la tanasa de A. kawachii IFO 4308. Debido a que no hay una estructura tridimensional reportada de una proteína con una alta identidad en su secuencia con la tanasa de A. niger GH1 que se encuentre disponible en el Protein Data Bank, fue necesario emplear el método threading para modelar la estructura tridimensional de la tanasa de A. niger GH1 a través del servidor Phyre2, uno de los mejores servidores según el “Critical Assessment of Protein Structure Prediction” (CASP) (Kelley y Sternberg, 2009). Con el modelo molecular se encontró que la tanasa tiene un plegamiento similar a la estructura tridimensional de una feruloilesterasa de A. oryzae cuyo código PDB es 3WMT (dos dominios estructurales: un dominio α/β-hidrolasa y un dominio de tapa), la cual tiene una identidad en la secuencia aminoacídica de 24% respecto a la tanasa de A. niger GH1. Adicionalmente, ambas estructuras tienen una triada catalítica que forma el dominio estructural CS-D-HC.
El presente trabajo es el primer reporte de una estructura molecular propuesta de una tanasa de la familia de las Tanasas PF07519 con un plegamiento muy similar a una feruloilesterasa, que tiene dos dominios estructurales y el motivo CS-D-HC. Sin embargo, la especificidad de las dos enzimas hacia su substrato es diferente, probablemente porque el sitio de unión al sustrato está formado por diferentes residuos de aminoácidos en las dos enzimas: Phe-225, Gln-228, Gln-229, Glu-365, Ser-279, Gly-381, e Ile-451 para la tanasa de A. niger GH1, y Phe-232, Leu-235, Thr-236, Tyr-348, Phe-354, Tyr-356, e Ile-419 para la feruloilesterasa de A. oryzae (Suzuki et al., 2014).
Actualmente ha sido reportada la producción de siete tanasas en microorganismos, Lactobacillus plan-tarum, Klebsiella pneumoniae, Bacillus licheniformis, Aspergillus awamori, Aspergillus oryzae y A. niger. Entre estas tanasas, seis fueron nativas y sólo una de A. oryzae fue producida como recombinante (Yu y Li, 2008; Zhong et al., 2004). De entre las tanasas nativas, cinco han sido producidas en cultivo sumergido convencional y sólo la de A. niger GH1 fue producida de manera extracelular en cultivo en estado sólido, siendo el nivel de producción más alto (2,291 U/L en 20 h) comparada a las otras tanasas nativas. Sin embargo, en el caso de la tanasa de A. niger GH1, se observó una disminución en la actividad después de 20 h de incubación, la cual estuvo asociada con un concomitante incremento de actividad de proteasas (Cruz et al., 2005).
La tanasa de A. oryzae ha sido producida en el sis-tema de expresión de P. pastoris de manera extracelular (Zhong et al., 2004) e intracelular (Yu y Li, 2008), resultando una actividad de 7 U/ml después de 96 h de inducción en un cultivo en lote alimentado empleando un biorreactor de 3 L en el primer caso, y 0.96 U/ml después de 72 h de inducción en un cultivo a nivel matraz para el segundo caso, ambos realizados con una cepa Muts. Sin embargo, en ningún caso se usaron genes sintéticos basados en el uso de codones preferenciales de P. pastoris y optimizado el contenido de AT para el gen de la tanasa o la secuencia codificante del péptido prepro del factor alfa de S. cerevisiae, ni se describieron las propiedades bioquímicas de las tanasas recombinantes, excepto por la formación de una estructura de doble cadena. Debido a que altas densidades celulares en P. pastoris pueden ser alcanzadas en un simple biorreactor en una forma controlada, se puede esperar un incremento de 10 a 100 veces en la producción de la tanasa de A. niger GH1 producida a nivel matraz, tal como se ha descrito para otras proteínas producidas en P. pastoris (Viader et al., 2013).
El servidor NetNGlyC 1.0 calculó once sitios potenciales de N-glicosilación para la tanasa de A. niger GH1. De entre estos sitios potenciales de N-glicosilación, tres (130NGSI, 265NLTS y 451NTTY) son más probables a ser N-glicosilados, ya que el modelo mo-lecular de la tanasa de A. niger GH1 mostró que estos tres sitios están en la superficie de la proteína. Además, los resultados del tratamiento con la glicosidasa endo Hf claramente demuestran que la tanasa recombinante fue N-glicosilada, ya que no se observó una banda en los geles de SDS-PAGE correspondiente la masa molecular teórica de la tanasa madura de A. niger GH1 (61.4 kDa), con base en la secuencia aminoacídica deducida a partir del gen de la tanasa. Sin embargo, se observaron principalmente dos bandas anchas (figura 2, carril 2) en masas moleculares menores (48.7 y 42.4 kDa) que la masa molecular teórica esperada para la tanasa madura de A. niger GH1, que se movieron a dos bandas definidas a masas moleculares de 34.6 y 30.3 kDa (figura 2, carril 3) después de la N-desglicosilación mediante la glicosidasa endo Hf. Estos resultados también indi-can que el concentrado enzimático de tanasa recombinante tuvo un alto grado de pureza. Además, la tanasa recombinante consistió de dos subunidades, probable-mente unidas mediante un puente disulfuro, generadas por un corte con la proteasa Kex2 del producto del gen de tanasa, ya que la secuencia de la tanasa madura de A. niger GH1 tiene dos pares de aminoácidos básicos (Lys-309-Arg-310, y Lys-347-Arg-348) que son un sitio típico de reconocimiento de la proteasa Kex2. Este tipo de estructura ha sido descrita para la tanasa nativa de A. oryzae (Hatamoto et al., 1996), y una tanasa de A. oryzae recombinante ya sea producida de forma extracelular (Zhong et al., 2004) o intracelular (Yu y Li, 2008, en P. pastoris. De los dos sitios de reconocimien-to Kex2, el sitio Lys-309-Arg-310 es más probable que sea reconocido por la proteasa Kex2, ya que el modelo molecular mostró que este sitio se encuentra en la superficie de la proteína en un loop flexible del dominio de tapa (figura 1C). Un corte por la proteasa Kex2 a un lado de Arg-310 generaría dos péptidos de 31.2 y 30.2 kDa. El puente disulfuro entre la Cys-195 y la Cys-495 uniría ambos péptidos.
Las tanasas de A. niger GH1 nativa y recombinante tienen la misma secuencia de aminoácidos. Por lo tanto, cualquier diferencia en sus propiedades bioquímicas (efecto del pH y temperatura en la actividad enzimática, y estabilidad) podría ser debido a sus diferentes grados de N-glicosilación o a sus diferentes estructuras, ya que la tanasa nativa de A. niger GH1 es principalmente una cadena glicopolipeptídica simple de 102-105 kDa (Renovato et al., 2011) y la tanasa recombinante es una proteína de doble cadena hiperglicosilada. La N-glico-silación afecta las propiedades bioquímicas como la masa molecular y el pH óptimo. Mientras que la tanasa nativa de A. niger GH1 mostró un pH óptimo de 6.0-7.0 y una temperatura óptima de 60ºC (Renovato et al., 2011), la tanasa de A. niger GH1 recombinante mostró un pH óptimo de 5.0 y una temperatura óptima de 20ºC. La tanasa recombinante producida en el presente traba-jo es la primera descrita del género Aspergillus con una temperatura óptima de 20ºC. Además, la tanasa de A. niger GH1 recombinante tuvo mayor actividad relativa en comparación con la nativa de A. niger GH1 en un intervalo de temperaturas de 10 a 40ºC, lo cual podría ser una ventaja para muchas aplicaciones en alimentos y bebidas (Chávez et al., 2012). Las tanasas nativa y recombinante mostraron alta actividad residual a 4ºC por al menos 120 h. Es de remarcar que las propiedades bioquímicas de una tanasa de Aspergillus con estructura de doble cadena no han sido descritas hasta hace poco (Mizuno, Shiono y Koseki, 2014). Sin embargo, en ese trabajo las propiedades bioquímicas de la tanasa nativa y recombinante fueron muy similares, probablemente porque ambas tuvieron una estructura de doble cadena, lo cual no es nuestro caso.
Basados en sus perfiles de actividad a diferentes valores de pH y temperatura, la tanasa producida podría ser utilizada en el procesado de alimentos y bebidas a bajas temperaturas.
Agradecimientos
Agradecemos al grupo de trabajo del Dr. Cristóbal Noé Aguilar González (Laboratorio de Alimentos, Facultad de Ciencias Químicas, Universidad Autónoma de Coahuila, Saltillo, México) por proporcionar el DNA genómico de A. niger GH1 y por sugerencias técnicas, así como a la Dra. Fabiola Veana por el apoyo técnico. J.A.F.G. agradece al Conacyt por la beca otorgada.
*Universidad Autónoma de Nuevo León.
Contacto: jose.viadersl@uanl.edu.mx
Referencias
Aguilar, C.N., Gutiérrez S., G. (2001). Review: sources, properties, applications and potential uses of tannin acyl hydrolase. Food Sci Technol Int 7, 373-382.
Chávez G., M., et al. (2012). Biotechnological advances and challenges of tannase: an overview. Food Bioprocess Technol. 5, 445-459.
Cruz H., M., et al. (2005). Isolation and evaluation of tannin-degrading fungal strains from the Mexican desert. Z. Naturforsch. C 60, 844-848.
Hatamoto, O., et al. (1996). Cloning and sequencing of the gene encoding tannase and a structural study of the tannase subunit from Aspergillus oryzae. Gene, 175, 215-221.
Kelley, L.A., Sternberg, M.J. (2009). Protein structure prediction on the Web: a case study using the Phyre server. Nat. Protoc. 4, 363-371.
Mizuno, T., Shiono, Y., Koseki, T. (2014). Biochemical characterization of Aspergillus oryzae native tanna-se and the recombinant enzyme expressed in Pichia pastoris. J. Biosci. Bioeng. 118, 392-395.
Renovato, J., et al. (2011). Differential properties of Aspergillus niger tannase produced under solid-state and submerged fermentations. Appl. Biochem. Bio-technol. 165, 382-395.
Sreekrishna, K. (2010). Pichia, optimization of protein expression. In: Flickinger M. C., editor. Encyclope-dia of industrial biotechnology: bioprocess, bioseparation, and cell technology. Hoboken: Wiley, pp. 1-16
Suzuki, K., et al. (2014). Crystal structure of a feruloyl esterase belonging to the tannase family: A disulfide bond near a catalytic triad. Proteins 82, 2857-2867.
Viader S., J.M., et al. (2013). Optimization of ¿ve environmental factors to increase beta-propeller phytase production in Pichia pastoris and impact on the physiological response of the host. Biotechnol. Prog. 29, 1377-1385.
Yu, X.W., Li, Y.Q. (2008). Expression of Aspergillus oryzae tannase in Pichia pastoris and its application in the synthesis of propyl gallate in organic solvent. Food Technol. Biotechnol. 46, 80-85.
Zhong, X., et al. (2004). Secretion, purification, and characterization of a recombinant Aspergillus oryzae tannase in Pichia pastoris. Protein Expr. Purif. 36, 165-169.
RECIBIDO: 18-08-2016
ACEPTADO: 05-09-2016
